臻话RNA | 第一期

近年来,肿瘤精准医疗飞速发展,尤其是ALK、ROS1、RET、NTRK、FGFR等以结构变异为主的靶向药物陆续上市,越来越多患者从中获益。目前,基于DNA变化分析为主的精准医疗正在被临床广泛应用,实际上,基于RNA的NGS检测(RNA-NGS)在结构变异检测上展现出更加优越的性能,已被《NCCN 非小细胞肺癌临床实践指南》、《NCCN 结肠癌临床实践指南》、《骨与软组织肿瘤二代测序中国专家共识》等指南及专家共识收录。同时,RNA种类繁多、功能复杂,RNA-NGS应用也更为广泛。肿瘤RNA知多少?本文将从RNA种类、检测方法对比、在肿瘤治疗临床实践中的广泛应用等方面切入,带您全面深刻了解RNA!
RNA种类
细胞中所能转录出的所有RNA的总和通称为转录组。按照是否编码蛋白质,RNA可分为:编码RNA(coding RNA)和非编码RNA(non-coding RNA, ncRNA)。其中,编码RNA主要指mRNA,占总RNA的3~7%[1],由DNA转录而成,包含制造蛋白质的遗传蓝图(图一),已被广泛研究;非编码RNA依据调控作用分为两类:管家非编码RNA(Housekeeping ncRNAs)和调节非编码RNA(Regulatory ncRNAs),主要包括lncRNA、circRNA、miRNA等,详见表一。
表一 常见非编码RNA种类及其特征[2,3,4]


图1.mRNA加工和出核[5]
管家ncRNA在细胞中大量表达,主要调节细胞一般功能;调节性ncRNA在表观遗传、转录和转录后水平上充当基因表达的调节剂[6]。在临床科研中各种调节性ncRNA已得到广泛的研究,如:lncRNA PCAT19,通过与HNRNPAB核糖核蛋白复合物的相互作用激活前列腺癌相关的多个基因[5];c-MYC 通过与 miR-17-92 启动子中的E-box元件结合来激活致癌 miR-17-92的转录[7];circ_100876高表达的食管癌患者生存时间明显缩短;circRNA_0023642是一种转移激活因子,它通过调节 EMT 信号通路促进细胞增殖、转移和侵袭等[8]。ncRNA在细胞中提供多种调节功能,这些RNA相互作用形成复杂的调控网络。

图2.ncRNA调控网络[2]
RNA检测方法对比
RNA功能错综复杂,对它的研究需基于可靠的检测方法,目前已有多种方法进行RNA检测,如:qRT-PCR、RNA测序(RNA-seq)、RNA panel检测、NanoString nCounter、DNA微阵列等等,不同检测方法各有所长,根据应用场景进行选择方为上策。
表二 不同RNA检测方法对比[9,10]

RNA-NGS已在肿瘤治疗临床实践中获得广泛应用
近年来,RNA-NGS已成为全转录组基因表达、mRNA剪接变异以及融合分析不可或缺的研究工具。随着NGS技术的发展,RNA-NGS已逐步从临床科研走向临床实践,使更多的患者获益。尤其是在基因融合、MET 14号外显子跳读变异等方面,RNA-NGS相较于DNA-NGS也具有较为显著的优势。
表三 不同融合检测方法对比

3.1 RNA-NGS 表现出优异的融合变异检测能力,扩大受益人群
基于RNA的融合检测可以弥补DNA检测层面由于内含子及复杂结构带来的问题。来自MSKCC研究的数据显示,使用 MSK-Fusion panel 对 MSK-IMPACT 检测为驱动基因突变阴性的232例肺癌患者RNA样本进行检测,仍有~14%的样本可通过RNA检测发现可用药的融合变异或MET 14外显子跳读变异[11]。
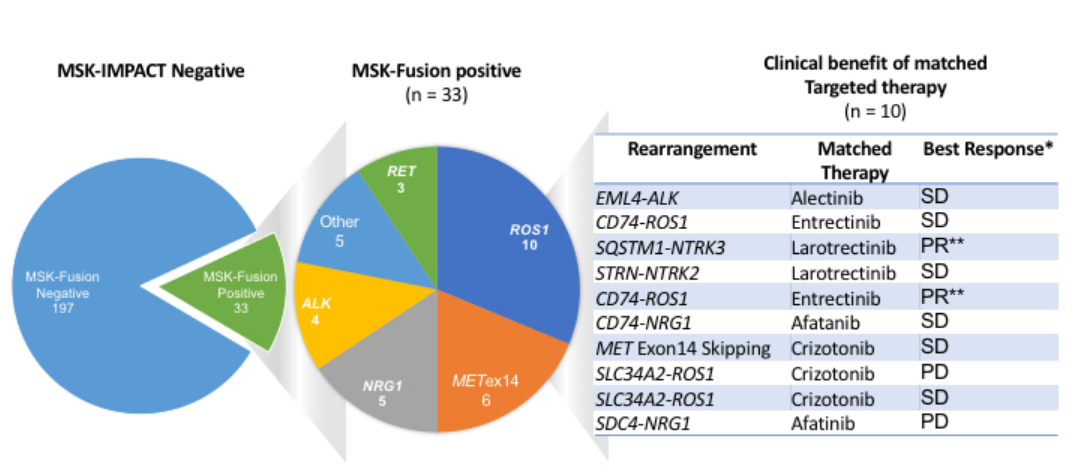
图3.基于RNA检测结果的患者匹配治疗情况
3.2 RNA-NGS测序助力精准分子分型
根据基因表达情况,进行膀胱癌分子分型、免疫分型(PD-L1、CD8A/CYT)分析,提示尤其是肌层浸润性膀胱癌(MIBC)等疾病相关的化疗、免疫治疗及预后[12],同时中华医学会泌尿外科学分会(CUA)、欧洲泌尿外科学会(EAU)指南也建议临床参考分子分型检测结果,判断预后,预测药物反应性,为膀胱癌精准诊疗提供参考。

图4.2020年发布在《欧洲泌尿学》杂志上的膀胱癌分子分型[11]
晚期肾癌患者的中位五年生存率低于10% ,FDA批准了多种血管内皮生长因子(VEGE)和mTOR抑制剂治疗晚期肾细胞癌。面对日益增多的治疗选择,需要更好地筛选人群以及了解肾癌的分子基础及耐药机制。根据RNA分子标志物已经在肾癌分子分型以及良恶性肿瘤区分方面取得了长足的进展,助力肾癌患者治疗方案的选择[13,14]。
3.3 基于RNA的检测在肿瘤免疫微环境领域持续发力
免疫治疗已在多种肿瘤中取得令人鼓舞的结果,基于PD-L1表达、TMB、MSI及免疫正负向相关基因等现有免疫疗效预测指标,仅能筛选部分患者获益于免疫治疗。免疫微环境作为最有潜力的生物标志物之一,已受到临床广泛的关注。探索与反应和耐药相关的肿瘤微环境将有助于更精确地识别从免疫治疗中获益的人群。
基于RNA检测,通过相关基因表达量计算T细胞炎性基因表达谱(GEP)评分,已在多项来自KEYNOTE系列临床试验数据中得到验证,用于评估肿瘤免疫微环境炎性程度,对免疫治疗响应者和未响应者进行分层[15,16]。
在接下来的几期文章中,小编将详细介绍关于RNA-NGS在肿瘤领域的临床应用及相关进展,敬请期待。