近日,求臻医学信息团队研发的一站式肿瘤精准诊疗知识挖掘平台OncoPubMiner,在国际权威生物信息学期刊Briefings in Bioinformatics(SCI影响因子:13.994,中科院JCR数学与计算生物学1区Top期刊)上在线发表 [1]。该平台利用自然语言处理技术(NLP,一种人工智能技术),对PubMed收录的2,343万篇摘要和PMC收录的437万篇全文进行了深入挖掘,识别出逾2.7亿实体-文献关系对。同时,平台创新性地设计了全流程一站式数据在线摘录功能,不仅为求臻医学实时追踪最新肿瘤学研究进展、快速更新肿瘤NGS检测结果解读知识库奠定了平台基础,也将为肿瘤患者的检测报告带来实时、全面、高质量的自动化解读。

研究背景
近年来,高通量测序技术(NGS)在精准医学领域发挥的作用日益凸显,在肿瘤精准医学领域更是方兴未艾,相关的研究论文数量也呈现指数式增长。作为开发新算法、新工具的金标准,高质量且实时更新的肿瘤精准诊疗知识库肩负着快速追踪科研进展,更好地服务于临床,辅助新研究发现的重要使命,更是实现自动化和智能化报告解读系统的基石。
诚然,现如今相关领域已有多款优秀数据库,如OncoKB [2]、CIViC[3]、JAX-CKB [4]、My Cancer Genome [5]、PMKB [6]、CGI [7]、OncoPDSS [8]等,但仍存在数据不全、缺乏持续或实时更新、数据无法批量获取等问题。因此,开发高质量且实时更新的自建知识库迫在眉睫。
研究设计
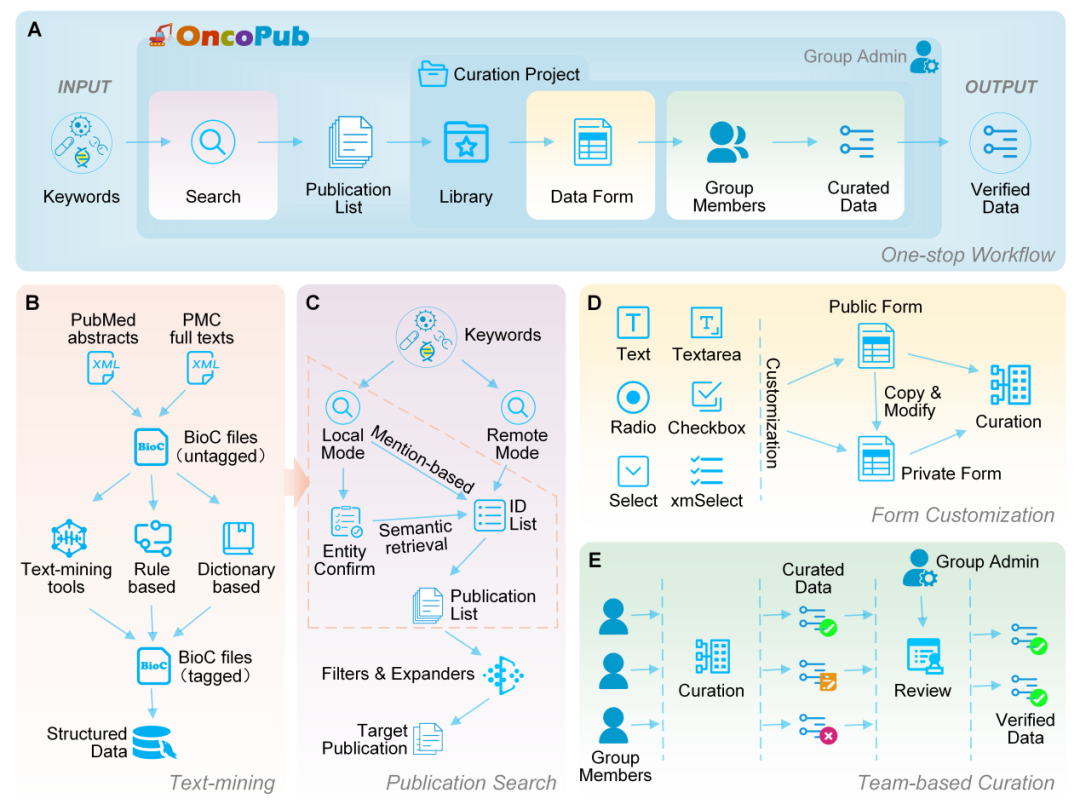
图1. OncoPubMiner平台工作流(workflow)
研究论文是科研发现的第一展示阵地,也是肿瘤精准诊疗知识数据的重要来源。本项目以文献数据挖掘为目标,从NCBI下载可公开使用(open-access)的PubMed摘要(Abstract)和PMC全文(Full-text),经过脚本处理、解析和格式转化后,利用基于人工智能的NLP技术对肿瘤精准诊疗相关的实体(如癌种、基因、变异、药物、临床意义、证据方向)进行挖掘。同时,利用业界常用的术语集(terminology)和本体(ontology),分别对自动化标注的实体进行标准化和层级化处理。最后,基于数据挖掘结果,构建一套包括文献检索、文库管理、团队搭建、表单定制、项目管理、数据摘录及审核等功能在内的全流程一站式平台。
主要研究结果
1.
文献挖掘
该工作首先从NCBI的FTP服务器下载全量的可公开使用的数据集。所下载的XML格式的文献数据均通过Python脚本转化为BioC-JSON(untagged,http://bioc.sourceforge.net/,图2),一种NCBI认可的生物医学文本处理与交互格式。

图2. BioC数据格式示例
接着,利用NLP技术,对全部转换后的BioC-JSON(untagged)数据进行实体识别(Entity Recognition)。现阶段识别的实体主要有:癌种/疾病、基因、变异/生物标志物、药物/化合物、临床意义和证据方向等。识别得到的实体再经过词典库(从公共数据库整理得到)和规则库(求臻医学信息团队文本挖掘经验总结得到)过滤,并经过同义词匹配和相似度计算等方式,对标注得到的全部实体进行标准化处理,从而产生终版的带有标准化标注结果的BioC-JSON(tagged)数据。文献挖掘已实现全流程自动化处理。
此外,该工作还开发了NCBI FTP服务器实时监控脚本OncoPubMonitor.py,一旦监控到有新文献释放(release),该脚本会自动进行增量下载,下载完成即启动自动化文本挖掘工作流。截至发稿,OncoPubMiner已挖掘23,434,007篇PubMed摘要、4,379,600篇PMC全文,识别出了277,198,162对去重后的“实体-文献”关系对。
2.
文献检索
文献挖掘结果分别以基于MongoDB的文档形式和基于MySQL的结构化形式进行存储。基于后者,该工作设计并开发了实体依赖的精准和模糊检索模式。此外,OncoPubMiner也集成了基于NCBI EUtilities API的远程检索模式。丰富的检索方式,能为用户带来更快更精准的文献检索体验。
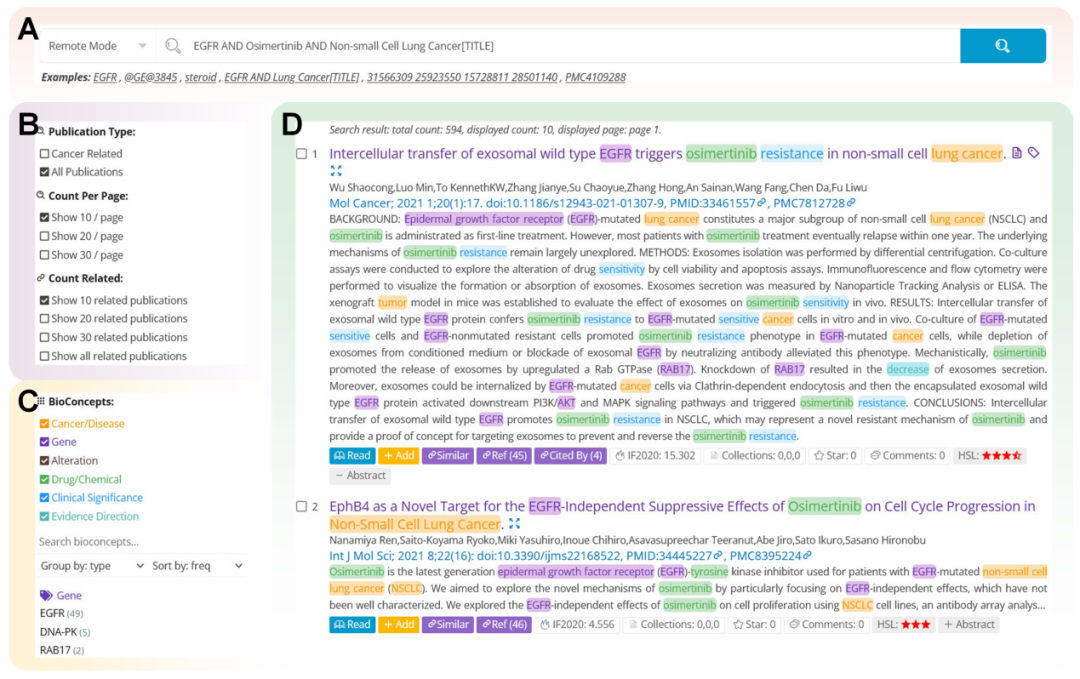
图3. OncoPubMiner平台文献检索功能
为便于用户更直观地筛选目标文献,OncoPubMiner还展示了文献最近两年的影响因子(IF2020和IF2021),计算了HSL(Highest-sentence level)分值,显示文库收录、标星以及备注的状态。不仅如此,该工作还实时统计关联了每一篇文献引用(Reference)、被引用(Cited_by)以及相似(Similar)的文献列表。通过关联检索,能利用初步锁定的“种子”文献更快速地发掘其他可能感兴趣的文章。
3.
表单定制
OncoPubMiner的设计初衷,既是为优化求臻医学的检测业务服务,也是为了整个肿瘤精准诊疗领域服务。不同的研究机构、企业,对于各自知识库的结构设计也有所差异,如何能让系统满足所有用户对于数据结构的需求,这是OncoPubMiner设计之初就在考虑的问题。求臻医学信息团队为OncoPubMiner集成了数据表单在线定制功能,所有用户均可根据实际需求,自定义数据采集表单,包括字段名称、类型、默认选项、是否必填/必选、排序位置、字段提示等,均可自行配置(图4)。
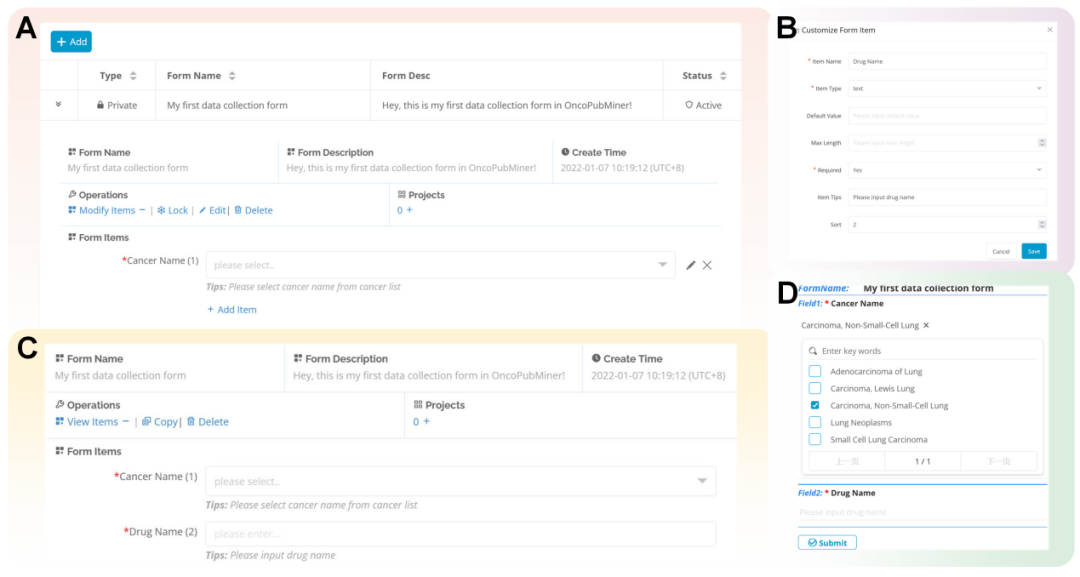
图4. OncoPubMiner表单自定义功能
值得一提的是,OncoPubMiner预定义了完全兼容CIViC数据平台的包括诊断(Diagnostic)、预测(Predictive)、预后(Prognostic)、功能(Functional)、易感性(Predisposing)、致癌性(Oncogenic)等6套数据采集表单。所有用户均可基于这些公共表单进行拷贝,并在此基础上进行修改,方便快速构建目标表单(图5)。
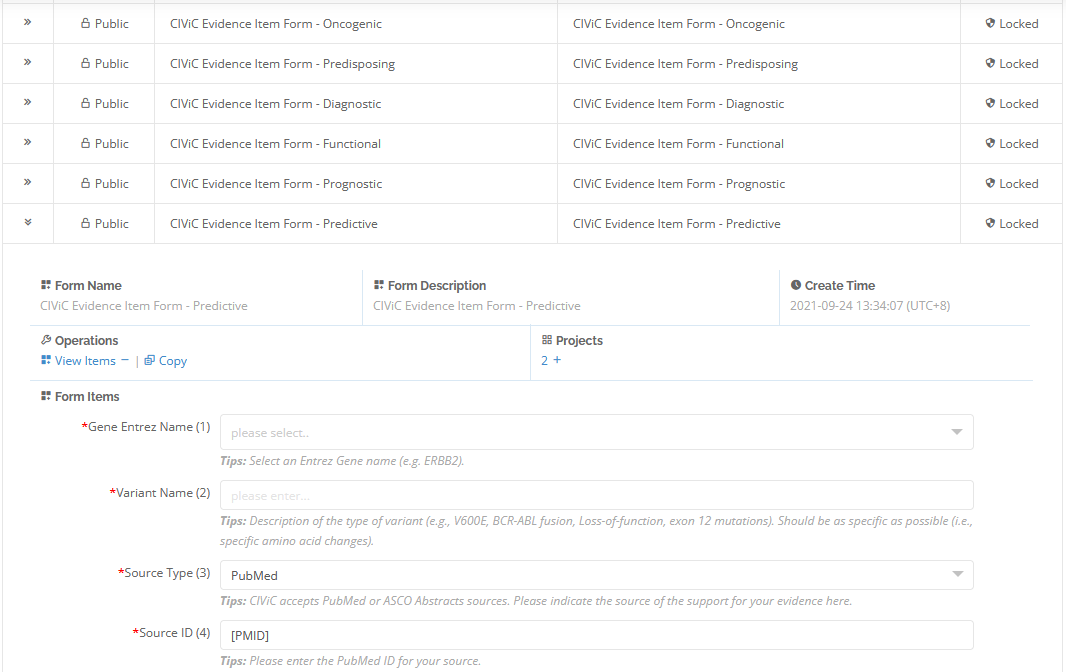
图5. OncoPubMiner预定义的兼容CIViC的数据采集表单
4.
项目管理
除了上述功能,OncoPubMiner还提供了文库、团队以及项目管理等功能。

图6. OncoPubMiner 文献阅读及数据采集平台
通过关键词检索得到的文献可以通过文库进行集中管理,同时用户可以创建团队成员账号,并建立数据摘录(data curation)项目,将文库、表单和团队成员添加到项目中来:以团队的形式对目标文献进行阅读,利用表单进行数据采集,并对所采集的数据进行审核(图6),最终得到高质量的结构化知识数据(图7)。

图7. OncoPubMiner导出的结构化知识数据
研究总结
本项目中,求臻医学研发了一套用于肿瘤精准诊疗文献挖掘的一站式平台。通过基于NLP技术的文本挖掘,构建了全面的肿瘤学知识挖掘数据库。基于该数据库,提供了丰富的文献检索和筛选功能,并设计了全流程文献数据摘录与审核功能。通过该平台,用户能够实现“关键词进,知识库出”,一站式构建高质量、及时更新、结构化的自建肿瘤精准诊疗知识库,彻底解决利用公共数据库时所遇到的质量不高、数据不全、更新不及时、无法商用等痛点问题。
求臻医学信息平台
求臻医学信息团队具备国际一流的研发水平,在生物信息分析、数据挖掘算法和平台开发方面经验丰富。在临床转化应用方面,信息团队搭建起了完备的肿瘤精准诊疗信息化平台,涵盖了样本信息管理ChosenCRM系统,实验室信息管理ChosenLIMS系统,全自动生信分析流程ChosenPipelines,肿瘤精准诊疗知识库ChosenMedKB,以及报告自动化信息管理ChosenReportInfo系统,实现了完整的业务信息流闭环。在此基础上,信息团队精心研发,不断打磨,在信息化、自动化、智能化上不断发力,全方位助力肿瘤精准医疗事业的蓬勃发展。
求臻医学信息平台官网
https://chosenmedinfo.com/
OncoPubMiner官网
https://oncopubminer.chosenmedinfo.com/
点击下方阅读原文查看文章






