近日,求臻医学首席科学家李蔚教授开创性地提出,位于基因的3′非翻译区(3′UTR)的选择性多聚腺苷酸化(APA)关联的遗传变异位点(3′aQTL)可以解释约16.1%的人类复杂疾病和性状差异,这将为解读功能性非编码区遗传变异(SNP)提供重要理论依据。该项研究对3′aQTL遗传变异的分子机理和与复杂疾病的关联进行了系统分析,为揭示人类复杂疾病的分子机制发挥了重要作用。目前,相关研究成果已在线发表于国际顶级学术期刊Nature Genetics(影响因子:38.33)。

研 究 背 景
尽管全基因组关联研究(GWAS)已经发现了数千种与数量性状和常见疾病相关的SNP位点。然而,绝大多数变异(近90%以上)位于非编码区,阐明这些变异的分子机制十分困难。传统上,研究人员常用表达数量性状(eQTLs)或可变剪切数量性状(sQTLs)来解释分子机制,虽然少量非编码SNP确实与相邻基因表达相关,但大部分与性状相关的非编码SNP的功能尚不清楚。
选择性多聚腺苷酸化(APA)是一种重要的前体RNA加工机制,广泛存在于所有真核生物中。通过在RNA 3′UTR不同位置上添加polyA尾巴,可以选择性地调节3′UTR的长短。由于3′UTR含有多种顺式调控元件,如:miRNA或RNA结合蛋白(RBP)结合位点,因此,APA可以通过调控3′UTR的长度,影响目标mRNA的稳定性和翻译效率以及翻译后蛋白质的细胞定位,进而精细调节基因表达,对一系列细胞过程(如增殖、分化和肿瘤发生)产生根本性的影响。

图1 APA通过调节3′UTR长度调控基因表达
虽然APA发生在大约70%的人类基因中,但是缺乏APA事件与疾病风险和复杂的人类性状表型之间的广泛关联分析,并且非编码SNP在不同人体组织中与APA及其表型特征和疾病的关系有待揭示。
研究方法与结果
为了系统研究非编码SNP在不同人体组织中与APA及其表型特征和疾病的关系,李蔚教授团队应用DaParsV2.0算法,基于来自467个个体,46种组织的8,722套RNA-seq与配套全基因测序数据,绘制非编码SNP与APA事件关联图谱。共鉴定到40万余个遗传变异与11,613个基因的APA事件相关,占所有已注释基因的51%。
这些与APA相关的数量性状被定义为3′QTLs。这些鉴定到的相关基因,一些已被前期的研究阐明,但绝大多数基因与3′QTLs的关系未被报道,其中不乏一些具有重要意义的基因,如在神经发育中起重要作用的CHURC1基因;与过敏性肠综合征相关的TPSAB1基因等。研究发现,3′QTLs可以解释平均25.2%的APA变异,16.2%的APA基因表达改变。

图2 3′QTLs与3′UTR长短相关
接下来,团队运用多元自适应收缩(MASH)评估了3′QTLs的组织分布。根据聚类结果,可以将46种组织划为脑组织与非脑组织。这些模式揭示了由于APA的调节形成不同组织间发育和功能的相似性。此外还发现,尽管78.4%的组织间3′aQTLs具有相同的位点特征,但只有13.9%的3′aQTLs表现出相似的程度特征。而85%组织间eQTLs具有相同位点特征,36%表现出相似的程度特征。这表明3′aQTLs比eQTLs具有更强的组织特异性。
为了进一步探究3′aQTLs分子表型机制,团队从polyA模体、RNA二级结构和RNA结合蛋白(RBP)结合位点改变方面进行了分析。研究表明,一些3′aQTLs通过改变经典polyA信号序列(PAS)的方式影响APA。还有一些可以通过改变AU,GU和尿嘧啶富集元件的方式影响APA。分析riboSNitch 数据与3′aQTL关联,发现3′aQTLs与RNA二级结构变异具有强相关性。应用CLIP-seq鉴定到了73个RBP更倾向于结合含有3′aQTLs区域。其中发现一个肿瘤抑制因子LARP4,经过一系列实验验证发现LARP4参与了APA调节,并且3′aQTLs可以通过影响LARP4对其位点的结合影响APA。这些结果揭示了3′aQTLs的分子表型机制,并且提示3′aQTLs分析可以作为全新APA调节因子的发现工具。

图3.实验验证LARP4调节APA
研 究 转 化
最后,李蔚教授团队分析了3′aQTLs与疾病风险的关系。通过关联分析23种常见人类疾病与表型相关SNP和3′aQTLs,鉴定到11.5%的组织特异的性状中富含3′aQTL变异。尤其值得注意的是,3′aQTLs在解释与溃疡性结肠炎、原发性胆管炎和阿尔茨海默病等多种自身免疫性疾病相关的遗传性方面尤其有效。
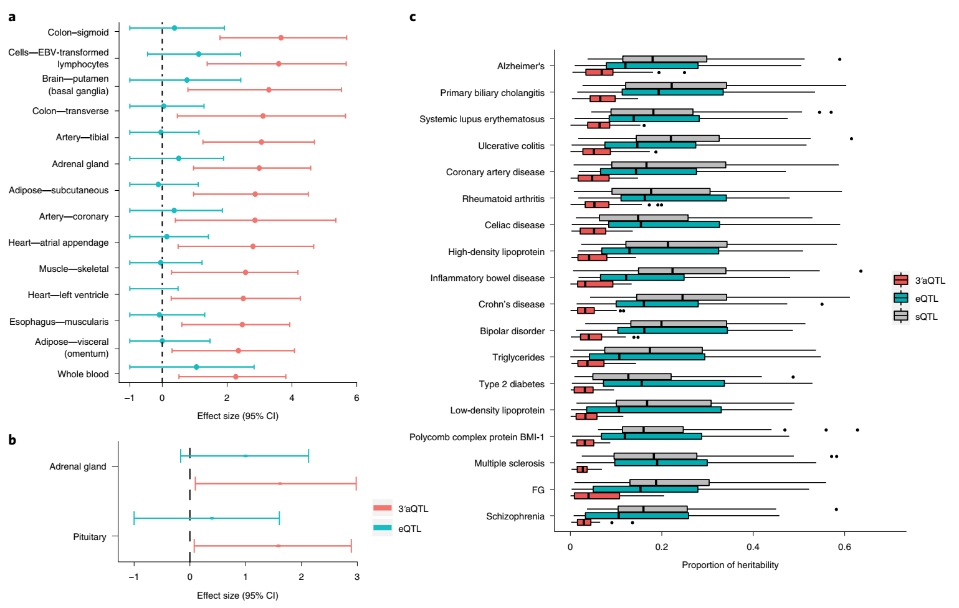
图4. 3′aQTL与疾病风险SNP的关系
DaPars鉴定到的3′QTLs适用于与遗传相关的绝大多数疾病,不仅可以作为生物标志物预测疾病风险,并且由于具有组织特异性,3′QTLs相关基因可以作为潜在的药物靶点,实现逆向转化研究,即从人群大数据出发——发现靶点——体外验证——应用于临床。本研究从组织水平证明了,APA对于细胞正常生理过程及多种重大遗传相关疾病发生发展的重要作用,该项工作对解释人类复杂疾病风险易感位点有着重要的推动作用,为揭示人类复杂性状和疾病病因学提供了新的方向。
与此同时,求臻医学首席科学家李蔚教授,带领团队从单细胞水平,证明了APA对于细胞正常生理过程及疾病发生发展的重要作用,敬请期待下一篇精彩成果!
李蔚教授团队,多年来专注APA领域基础研究,
在过去几年里做出了一系列原创性的发现:
2014年,李蔚教授团队开发了首个从传统RNA-seq数据里直接分析APA的生物信息算法DaPars(Nature Communications 2014);同年发现了CFIm25蛋白通过广泛调控APA,抑制胶质母细胞瘤发生发展(Nature 2014) 。
2) 2018年, 李蔚教授团队报道了在乳腺癌中APA调控的3′UTR缩短(Nature Genetics 2018),破坏了竞争性内源RNA的信号交互,导致了抑癌基因的反式抑制效应。
3) 2020年,李蔚教授团队发现肿瘤异常表达MAGE-A11泛素连接酶,干扰CFIm25对于APA的调控作用,导致肿瘤细胞广泛的3′UTR缩短,调节紊乱,为理解肿瘤形成提供了新的证据(Molecular Cell2020)。
李蔚教授
求臻医学联合创始人&首席科学家
美国加州大学(尔湾)终身教授,讲席教授
主要研究方向:设计和应用生物信息学算法来评估肿瘤等疾病发生发展过程中的全局调控机制,在大规模基因组数据及表观遗传学数据分析方面有着坚实的基础。参与完成人类基因组计划,主导完成了中国第一个微生物基因组计划等重大项目。所开发的BSMAP、RSeqQC等算法,目前已成为Bisulfite-seq、RNA-seq数据分析等多个生信领域的标准算法。现已在高影响因子期刊上发表超过190篇论文,其中23篇作为通讯作者发表在Nature、Science、Cell系列刊物(包括子刊),H-index 高达80(发表的论文中有80篇引用超过80次)。
求臻医学首席科学家李蔚教授研究团队
诚招博士后2名
研究方向
通过表观遗传的大规模数据挖掘来解释肿瘤等人类复杂疾病
岗位要求
1.获得与生物信息学相关的博士学位;
2.具有分子生物学、肿瘤学研究背景,从事过肿瘤表观遗传学工作的人员优先;
3.博士期间,以第一作者发表过涉及生物信息分析内容的SCI论文;
4.具有良好的团队合作与沟通能力,以及较强的中英文写作能力。
工作地点
求臻医学科技(北京)有限公司或美国加州大学
简历投递
Wei.li@uci.edu
目前,李蔚教授已有6位学生在美国一流研究型大学(包含哈佛医学院、匹兹堡大学和梅奥诊所),获得独立PI的教授职位,2位学生已获得中国国家青年千人学术头衔。详情可参见:https://sites.uci.edu/weililab/。







