近日,求臻医学科研团队基于自身专业的基因组信息分析能力,结合对肿瘤基因组图谱(TCGA)数据库中II/III期结直肠癌(CRC)患者队列的综合分析,助力合作伙伴创新性地提出了新型免疫预后甲基化标志物模型—CG Score 。该模型能有效预测II-III期CRC患者免疫治疗疗效,将为改善CRC患者免疫治疗预后,带来新的临床获益。
目前,相关科研成果已在线发表于学术期刊 Frontiers in Genetics(影响因子:4.599),论文题目为“Identification of a Novel Immune-Related CpG Methylation Signature to Predict Prognosis in Stage II/III Colorectal Cancer”。
我国CRC发病率已位居恶性肿瘤发病率第五位,大约70% 的CRC患者发现时已处于II/III 期。目前,TNM分期标准在CRC患者预测预后及做出临床决策方面存在不足,尤其是在II/III 期CRC患者中。
随着临床对免疫治疗机制的深入理解,肿瘤免疫治疗虽已取得长足的进步,但CRC患者从免疫治疗中获益有限,临床研究进展相对滞后。此外,现有的生物标志物不能有效预测MSS CRC患者的预后。因此,迫切需要挖掘预测CRC患者预后的免疫相关生物标志物,以提高结直肠癌免疫治疗疗效。
研究发现,异常的DNA甲基化导致基因表达下调,并可能诱发癌症,而且有潜力成为肿瘤生物标志物。然而,只有少数研究集中于识别免疫相关甲基化特征,以预测II/III期结直肠癌患者的预后。因此,有必要确定预后相关甲基化生物标志物。
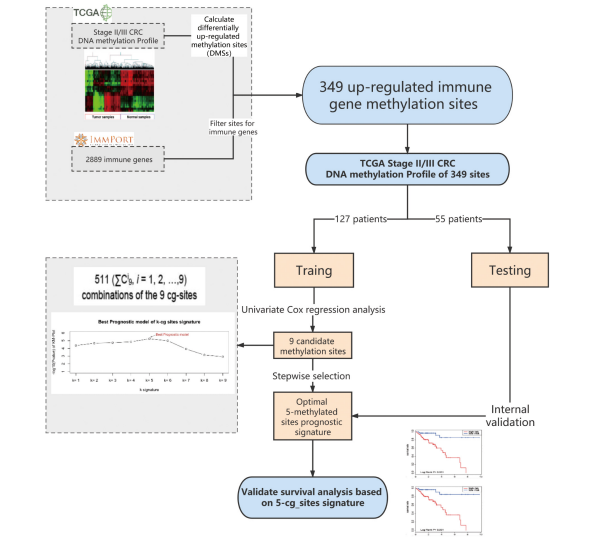
本研究旨在运用基因组信息分析方法,对肿瘤基因组图谱(TCGA)数据库中CRC队列的甲基化芯片数据和肿瘤免疫相关基因(InnateDB; ImmPort)进行了综合分析。通过逐步回归相关迭代性算法,确定5个免疫相关的差异甲基化位点,继而通过lasso-cox回归分析方法构建免疫风险甲基化特征预后模型(CG Score)。最后,在独立数据集上评估CG Score的预后效能,并进一步探讨免疫细胞浸润与免疫治疗生物标志物的相关性。
1.免疫风险预后模型CG Score的构建及预后效能评估
本研究通过逐步回归相关迭代性算法,确定了5个免疫相关的差异甲基化位点(cg11621464、cg13565656、cg18976437、cg20505223和cg20528583,继而通过lasso-cox回归分析方法构建了免疫风险甲基化特征预后模型(CG Score)(图1)。
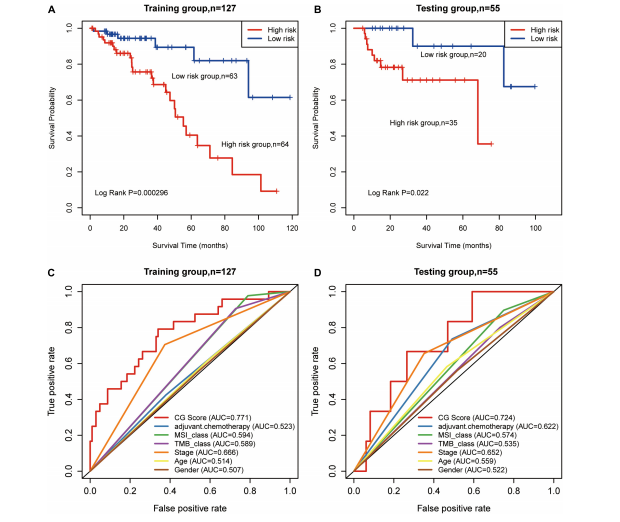
根据CG Score将患者分为高危组和低危组,在训练集(图2 A)和检验集(图2 B)中,预后模型对5年生存期的预测曲线下面积(Area Under the Curve, AUC)分别为0.771和0.724(图C和图D),高危组样本比低危组样本的总生存期均更短(Log Rank p<0.05)。
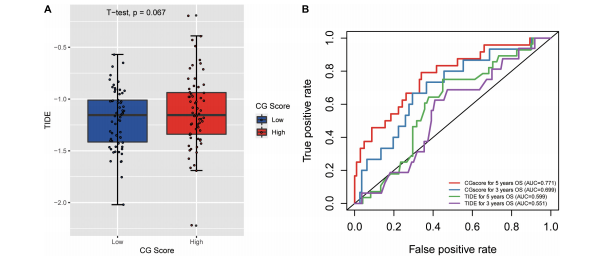
肿瘤免疫功能失调和排斥(TIDE)是一种现有基于基因表达的免疫治疗预测生物标志物,旨在预测对免疫检查点封锁(ICB)治疗的临床反应。本研究中,CG Score显示出与TIDE-score 的显著正相关性(图3A)。另外在训练集队列中,CG Score模型对于预测CRC患者免疫治疗的反应依旧优势凸显(图3B)。模型的3年和5年预后AUC分别为0.699和0.771。
图3.评估CG Score对ICB治疗反应的预测效能
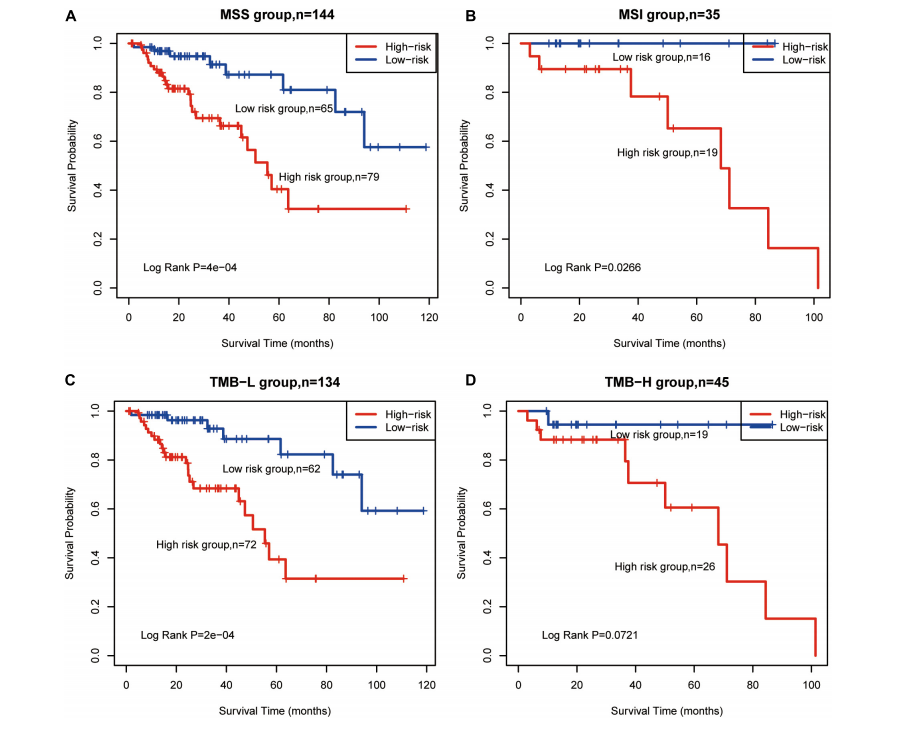
2. 探索CG Score对于特定亚型人群的预后效能
文章阐述了在特定的亚型人群中,如MSS(图4A)、MSI(图4B)、TMB-Low(图4C)和TMB-High (图4D),根据CG Score将患者分为高危组和低危组。结果显示,高危组样本比低危组样本的总生存期均更短(Log Rank p<0.05),该模型为预测CRC患者免疫治疗疗效评估(或预测)提供了潜在的生物标志物。
本研究确立了基于5个免疫相关甲基化位点的风险预后评分模型——CG Score,该模型能有效反应CRC患者的免疫应答水平,对改善CRC患者的预后具有重要临床指导意义。此外,研究进一步发现,CG Score 可以区分MSS、MSI、TMB-L和TMB-H亚型患者的预后,有可能成为一种新的免疫相关预后生物标志物,助力结直肠癌患者的预后评估。
参考文献:
1. Antelo, M., Balaguer, F., Shia, J., Shen, Y., Hur, K., Moreira, L., et al. (2012). A high degree of LINE-1 hypomethylation is a unique feature of early onset colorectal cancer. PLoS One 7:e45357. doi: 10.1371/journal.pone.0045357
2. Bathe, O. F., and Farshidfar, F. (2014). From genotype to functional phenotype: unraveling the metabolomic features of colorectal cancer. Genes (Basel) 5, 536–560. doi: 10.3390/genes5030536
3. Berg, M., Agesen, T. H., Thiis-Evensen, E., INFAC-study group, Merok, M. A.,Teixeira, M. R., et al. (2010). Distinct high resolution genome profiles of early onset and late onset colorectal cancer integrated with gene expression data identify candidate susceptibility loci. Mol. Cancer 9:100. doi: 10.1186/1476-4598-9-100
4. Chen, J., Gingold, J. A., and Su, X. (2019). Immunomodulatory TGF-β signaling in hepatocellular carcinoma. Trends Mol. Med. 25, 1010–1023. doi: 10.1016/j.molmed.2019.06.007
5. Chen, J.-S., Huang, J.-Q., Luo, B., Dong, S.-H., Wang, R.-C., Jiang, Z.-K., et al. (2019). PIK3CD induces cell growth and invasion by activating AKT/GSK-3β/β-catenin signaling in colorectal cancer. Cancer Sci. 110, 997–1011. doi:10.1111/cas.13931
6. Chen, W. (2015). Cancer statistics: updated cancer burden in China. Chin. J. Cancer Res. 27:1. doi: 10.3978/j.issn.1000-9604.2015.02.07
7. Edge, S. B., and Compton, C. C. (2010). The American Joint Committee on Cancer: the 7th edition of the AJCC cancer staging manual and the future of TNM. Ann. Surg. Oncol. 17, 1471–1474. doi: 10.1245/s10434-010-0985-4
8. Fang, J.-Y., Dong, H.-L., Sang, X.-J., Xie, B., Wu, K.-S., Du, P.-L., et al. (2015). Colorectal cancer mortality characteristics and predictions in China, 1991–2011. Asian Pac. J. Cancer Prev. 7991–7995. doi: 10.7314/apjcp.2015.16.17.7991
9. Guo, J.-C., Wu, Y., Chen, Y., Pan, F., Wu, Z.-Y., Zhang, J.-S., et al. (2018). Proteincoding genes combined with long noncoding RNA as a novel transcriptome molecular staging model to predict the survival of patients with esophageal squamous cell carcinoma. Cancer Commun (Lond). 38: 4. doi: 10.1186/s40880-018-0277-0