近日,求臻医学科研团队自主设计并实现了一套可定制、一体化肿瘤突变检测软件系统——DIVIS(variants Detection、Interpretation、Visualization and Infrastructures)。该软件系统能够准确检测WGS、WES、Gene-Panel Sequencing的体细胞突变和种系变异,同时支持图形化的快速定制和分布式并行计算,是快速开展肿瘤基因组大数据挖掘的“利器”。目前,该研究成果已在国际肿瘤学领域重要科技期刊Frontiers in Oncology(SCI 影响因子:4.848,中科院JCR 2区)上在线发表。

|Download|
■ DIVIS代码 :
https://github.com/niu-lab/DIVIS
■ docker镜像:
https://hub.docker.com/repository/docker/sunshinerain/divis
研究背景
目前,癌症基因组数据分析面临三个主要问题:
1. 突变识别软件一致性低导致体细胞突变假阳性。随着测序数据量的超摩尔定律增长和测序价格的不断下降,突变识别算法也层出不穷,这给突变识别算法选择及组合造成一定的困难,进而导致体细胞突变假阳性率高。
2.分析流程多样化,无法满足多种应用场景。不同的测序类型(WGS、WES、Gene-Panel)、样本组合(肿瘤-正常配对样本、单肿瘤组织样本,ctDNA等)、测序特征(测序区间、测序深度等)等,在特定需求(如超低突变频率检测)下产生了多样的流程。
3.分析流程的定制和维护复杂。现有的流程化软件多基于脚本语言或工作流描述语言,按需定制和更新维护都需要大量的编程,导致科研和生产效率低下。
因此,设计实现分布式、轻量级、图形化通用流程定制软件系统,开发面向多数据类型、多样本组合和多应用需求的一体化分析工具,快速、准确地定位致病位点,具有巨大的科研和生产价值。
研究设计
求臻医学科研团队首先研发了分布式轻量级通用工作流系统GPyFlow,如图1所示,GPyFlow主要由三个独立的模块GPyFlow-Visual、GPyFlow-CLI和GPyFlow-Workflow组成。
GPyFlow-Visual是前端交互式图形操作用户界面,通过单击、拖拽和连线等简单操作功能,可实现快速创建分析流程。GPyFlow-CLI是单机自动化流程执行器,将GPyFlow-Visual导出的JSON格式的流程规范构建为有向无环图,然后利用拓扑排序算法渲染出实际要运行的命令,以GPyFlow-CLI为底层程序而开发的GPyFlow-MPI通过MPI计算框架实现节点通信而完成多样本多流程的并行计算分析。
GPyFlow-Workflows是工作流归档、复用、共享平台,与GPyFLow-Visual、GPyFLow-CLI的联动可以实现对已有生信流程的再编辑。

图1. GPyFlow框架图
DIVIS主要功能包括肿瘤基因组数据分析的四阶段操作:下机数据预处理、序列比对和校正、突变识别、突变注释和质量控制等。如图2所示,为了灵活应用于各种分析场景,DIVIS分为流程式(pipeline)模块和子功能(substep)模块。流程式模块指从原始下机预处理到突变注释的完整分析模块。而子功能模块实现整个流程中的某个子过程,例如只进行比对或者进行从比对到突变注释的过程等。为了解决在网络隔离的环境下部分软件编译安装困难的问题,DIVIS同时发布了Docker镜像。
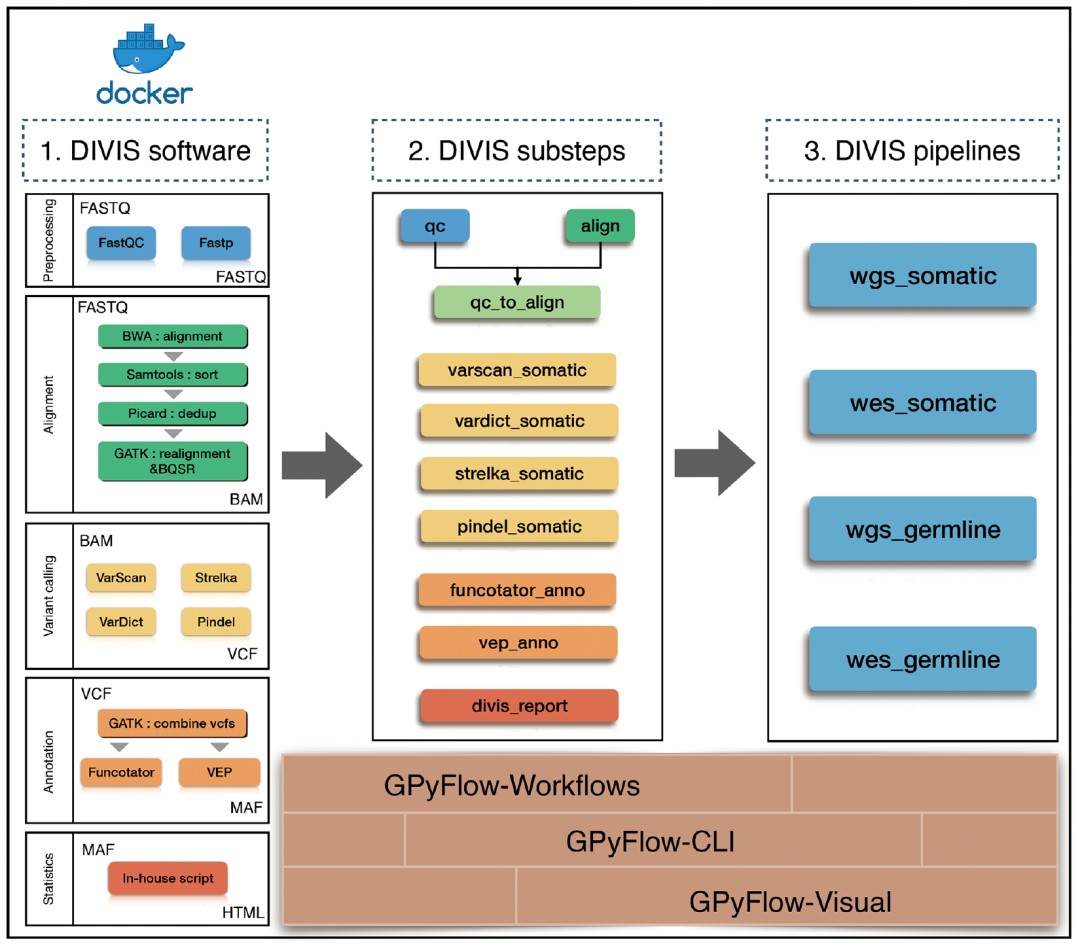
图2.DIVIS的整体架构
研究结果
另外,DIVIS亦可实现图形化的报告输出。该报告对应于肿瘤基因组数据分析的四个阶段,包含原始数据质控、比对数据质控、突变统计(图3)和癌基因注释等。

图3.基因突变结果统计
DIVIS中对体细胞突变的识别基于算法互补策略,SNVs结果来源于等位基因频率分析算法Strelka2和启发式阈值分析算法VarScan2、VarDict,并从中筛选至少2/3算法支持的位点,同理,indels的识别也采用三种不同算法(Pindel、Strelka2、VarScan2和VarDict)并从中筛选至少1/2支持的位点。算法互补策略的优势在于能够最大限度的避免漏检,尤其是对不同VAF的突变位点。在算法互补策略之上,设置筛选比例保证了位点的置信度,每个位点具有50%的算法支持率,克服了单个检测算法的弊端。
DIVIS能够准确识别体细胞突变,实现肿瘤突变检测可定制分析,可极大提高科研生产效率!与此同时,DIVIS有助于临床分析耐药机制和制定耐药应对策略,更好的评估治疗疗效并制定个体化精准治疗方案,进而为肿瘤患者争取更好的临床获益。
生信算法开发能力国际领先
求臻医学具备国际顶尖的基因组信息学团队,拥有国际领先的生物信息分析平台及算法,所开发的MACS、MSIsensor、HotSpot3D等算法已在业内应用并广受好评。例如,求臻医学自主研发的MSIsensor算法被FDA批准的首个基于NGS的肿瘤多基因检测试剂盒(MSK-IMPACT)所采用,同时被近期发布的《结直肠癌分子检测高通量测序中国专家共识》所推荐。
在MSIsensor的基础上,求臻医学多环节引入人工智能技术,持续研发了基于单肿瘤组织和外周血游离DNA(cfDNA)测序数据的MSI状态检测算法MSIsensor2与MSIsensor-ct。
目前,MSIsensor/MSIsensor2/MSIsensor-ct系列算法已经被超过310篇论文引用,为国际上引用数最高的泛癌种NGS-MSI探测工具集。
求臻医学与国内外科研团队合作,成功研发了一系列拥有自主知识产权的分析软件,例如用于RNA-seq数据分析的RseQC,用于甲基化分析的BSMAP、MOABS、Canyons,用于显著突变基因识别的MuSiC2等。尤其在检测免疫治疗关键生物标志物TMB和MSI的核心算法方面,求臻医学独具优势。
参考文献:
1、Martínez-Jiménez F, Muiños F, Sentís I, Deu-Pons J, Reyes-Salazar I, Arnedo-Pac C, et al. A compendium of mutational cancer driver genes. Nat Rev Cancer (2020) 20(10):555-72. doi: 10.1038/s41568-020-0290-x.
2、 Yurgelun MB, Chittenden AB, Morales-Oyarvide V, Rubinson DA, Dunne RF, Kozak MM, et al. Germline cancer susceptibility gene variants, somatic second hits, and survival outcomes in patients with resected pancreatic cancer. Genet Med (2019) 21(1):213-23. doi: 10.1038/s41436-018-0009-5.
3、Dietlein F, Weghorn D, Taylor-Weiner A, Richters A, Reardon B, Liu D, et al. Identification of cancer driver genes based on nucleotide context. Nat Genet (2020) 52(2):208-18. doi: 10.1038/s41588-019-0572-y.
4、He X, Chen S, Li R, Han X, He Z, Yuan D, et al. Comprehensive fundamental somatic variant calling and quality management strategies for human cancer genomes. Brief Bioinform (2020) 22(3). doi: 10.1093/bib/bbaa083.






