近日,求臻医学首席科学家李蔚教授首创性开发了一种可分析标准单细胞RNA测序数据(scRNA-seq) 多聚腺苷酸化(APA)事件的算法——scDaPars。scDaPars算法在DaPars算法基础上,有能力识别传统基因表达分析所不能区分的细胞亚群;采用了一种回归模型,能够在相关细胞之间分享APA信息;在处理有噪声的scRNA-seq数据时,解决了相关序列稀少的问题,在细胞亚群鉴定方面具有相当强的抗干扰能力(鲁棒性)。目前,该文章在线发表于国际重要科技期刊Genome Research(影响因子:9.043)上。
本研究首先测试了算法识别单细胞中近端polyA位点的能力。scDaPars可以在scRNA-seq数据中预测出84% bulk数据中的polyA位点;并且66.2%的预测位点与数据库中注释位点一致。通过模体分析,在预测polyA位点上游100 bp内鉴定出经典PAS序列,相关度P=1.2×10-44。表明scDaPars在预测polyA位点时具有高度准确性。
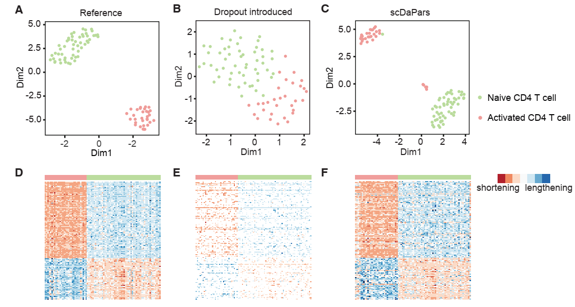
接下来利用模拟数据进行数据截取,在有效数据量下降的情况下,scDaPars依然可以将细胞亚群清晰的分开,即使数据量降低70%的情况下,分类AUC大于0.75,显示出scDaPars识别APA事件的强大能力。
将scDaPars分析真实乳腺癌Smart-seq2单细胞数据,发现与正常细胞相比,肿瘤细胞3’UTR显著缩短,并且依靠APA事件可以将肿瘤与非肿瘤细胞明显分开。肿瘤细胞不仅被scDaPars分到患者特异性簇中,还进一步被分为不同的分子亚群,证明了肿瘤间和肿瘤内亚型间存在APA异质性。不同的是,非肿瘤细胞主要根据其细胞类型(B细胞、髓细胞和T细胞)而不是患者进行聚集。
这一结果不仅证实了动态APA事件是免疫细胞的细胞类型特异性特征,而且表明在肿瘤细胞中观察到的患者特异性APA谱不太可能是由于患者样本中的批次效应造成的,而是反映了真实的肿瘤间APA变异。不仅如此,与先验知识一致的是算法可以将B细胞分为两个亚群,而只应用基因表达算法无法完成亚群的鉴定。说明scDaPars可以改善单细胞亚群分类的效果。
APA模式与细胞分化高度相关,为了验证scDaPars算法是否可以鉴定到仅靠基因表达谱无法区分的新细胞亚群,求臻医学科研团队分析了人内胚层发育6个不同时间点的758个单细胞测序数据。scDaPars算法的确可以鉴别出新的细胞亚群,例如,分化96小时的细胞被分为两个以前未被确认的亚群——通过分析两个亚群之间的APA和基因表达,发现单独使用APA可以准确地将两个亚群分开,然而,仅使用基因表达谱无法区分这两个亚群,表明scDaPars算法的优越性。
scDaPars使我们能够在转录后APA水平上了解细胞异质性,并且来自单细胞的APA信息,有助于识别传统基因表达分析所不能区分的细胞亚群。求臻医学首席科学家李蔚教授发表的两篇科研文章(上一篇Nature Genetics),分别从不同水平不同角度证明了APA对于细胞正常生理过程及疾病发生发展的重要作用。而以APA为表型的3‘QTLs适用于与遗传相关的绝大多数疾病。
李蔚教授表示:通过3’QTLs分析已经成功预测到一个与前列腺癌发生高度相关的驱动基因,抑制该基因阻碍肿瘤进展的同时不影响正常组织,有望成为理想的用药靶点,相关研究成果将在不久之后将与大家见面。与此同时,求臻医学将利用3’QTLs分析技术,推动自身对于疾病风险预测的理解与解读能力的进步,并助力发现全新疾病相关靶点,为后期药企合作奠定基础。
李蔚教授团队,多年来专注APA领域基础研究,在过去几年里做出了一系列原创性的发现:
2014年,李蔚教授团队开发了首个从传统RNA-seq数据里直接分析APA的生物信息算法DaPars(Nature Communications 2014);同年发现了CFIm25蛋白通过广泛调控APA,抑制胶质母细胞瘤发生发展(Nature 2014) 。
2) 2018年, 李蔚教授团队报道了在乳腺癌中APA调控的3′UTR缩短(Nature Genetics 2018),破坏了竞争性内源RNA的信号交互,导致了抑癌基因的反式抑制效应。
3) 2020年,李蔚教授团队发现肿瘤异常表达MAGE-A11泛素连接酶,干扰CFIm25对于APA的调控作用,导致肿瘤细胞广泛的3′UTR缩短,调节紊乱,为理解肿瘤形成提供了新的证据(Molecular Cell2020)。
求臻医学联合创始人&首席科学家
美国加州大学(尔湾)终身教授,讲席教授
主要研究方向:设计和应用生物信息学算法来评估肿瘤等疾病发生发展过程中的全局调控机制,在大规模基因组数据及表观遗传学数据分析方面有着坚实的基础。参与完成人类基因组计划,主导完成了中国第一个微生物基因组计划等重大项目。所开发的BSMAP、RSeqQC等算法,目前已成为Bisulfite-seq、RNA-seq数据分析等多个生信领域的标准算法。现已在高影响因子期刊上发表超过190篇论文,其中23篇作为通讯作者发表在Nature、Science、Cell系列刊物(包括子刊),H-index 高达80(发表的论文中有80篇引用超过80次)。
通过表观遗传的大规模数据挖掘来解释肿瘤等人类复杂疾病
1.获得与生物信息学相关的博士学位;
2.具有分子生物学、肿瘤学研究背景,从事过肿瘤表观遗传学工作的人员优先;
3.博士期间,以第一作者发表过涉及生物信息分析内容的SCI论文;
4.具有良好的团队合作与沟通能力,以及较强的中英文写作能力。
目前,李蔚教授已有6位学生在美国一流研究型大学(包含哈佛医学院、匹兹堡大学和梅奥诊所),获得独立PI的教授职位,2位学生已获得中国国家青年千人学术头衔。详情可参见:https://sites.uci.edu/weililab/。
参考文献:
1 Brennecke, P. et al. Accounting for technical noise in single-cell RNA-seq experiments. Nature methods 10, 1093-1095, doi:10.1038/nmeth.2645 (2013).2 Chu, L. F. et al. Single-cell RNA-seq reveals novel regulators of human embryonic stem cell differentiation to definitive endoderm. Genome biology 17, 173, doi:10.1186/s13059-016-1033-x (2016).3 Chung, W. et al. Single-cell RNA-seq enables comprehensive tumour and immune cell profiling in primary breast cancer. Nature communications 8, 15081, doi:10.1038/ncomms15081 (2017).4 Elkon, R., Ugalde, A. P. & Agami, R. Alternative cleavage and polyadenylation: extent, regulation and function. Nature reviews. Genetics 14, 496-506, doi:10.1038/nrg3482 (2013).5 Gao, Y., Li, L., Amos, C. I. & Li, W. Analysis of alternative polyadenylation from single-cell RNA-seq using scDaPars reveals cell subpopulations invisible to gene expression. Genome research, doi:10.1101/gr.271346.120 (2021).6 Gruber, A. J. & Zavolan, M. Alternative cleavage and polyadenylation in health and disease. Nature reviews. Genetics 20, 599-614, doi:10.1038/s41576-019-0145-z (2019).7 Kim, N., Chung, W., Eum, H. H., Lee, H. O. & Park, W. Y. Alternative polyadenylation of single cells delineates cell types and serves as a prognostic marker in early stage breast cancer. PloS one 14, e0217196, doi:10.1371/journal.pone.0217196 (2019).8 Li, L. et al. An atlas of alternative polyadenylation quantitative trait loci contributing to complex trait and disease heritability. Nature genetics, doi:10.1038/s41588-021-00864-5 (2021).9 Li, W. V. & Li, J. J. An accurate and robust imputation method scImpute for single-cell RNA-seq data. Nature communications 9, 997, doi:10.1038/s41467-018-03405-7 (2018).10 Sandberg, R., Neilson, J. R., Sarma, A., Sharp, P. A. & Burge, C. B. Proliferating cells express mRNAs with shortened 3' untranslated regions and fewer microRNA target sites. Science 320, 1643-1647, doi:10.1126/science.1155390 (2008).11 Shulman, E. D. & Elkon, R. Cell-type-specific analysis of alternative polyadenylation using single-cell transcriptomics data. Nucleic acids research 47, 10027-10039, doi:10.1093/nar/gkz781 (2019).12 Tian, B. & Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature reviews. Molecular cell biology 18, 18-30, doi:10.1038/nrm.2016.116 (2017).13 Velten, L. et al. Single-cell polyadenylation site mapping reveals 3' isoform choice variability. Molecular systems biology 11, 812, doi:10.15252/msb.20156198 (2015).14 Xia, Z. et al. Dynamic analyses of alternative polyadenylation from RNA-seq reveal a 3'-UTR landscape across seven tumour types. Nature communications 5, 5274, doi:10.1038/ncomms6274 (2014).15 Ye, C. et al. scDAPA: detection and visualization of dynamic alternative polyadenylation from single cell RNA-seq data. Bioinformatics 36, 1262-1264, doi:10.1093/bioinformatics/btz701 (2020).