求臻医学首席科学家李蔚教授首创性开发了一种基于大数据预测癌症驱动基因的算法——DORGE,通过全面整合遗传和表观遗传数据,DORGE算法可识别未知的肿瘤抑制基因(TSGs)和原癌基因(OGs)。这是首次尝试使用肿瘤基因变异以外的特征预测癌症驱动基因,既加深了对表观遗传学在肿瘤发生过程中机制的理解,又进一步彰显了新预测基因作为潜在治疗靶点的广阔前景。该成果已发表在国际重要科技期刊Science Advances (SCI影响因子:14.136)。

研 究 背 景
细胞内重要基因变异的累积会导致癌症,这些变异破坏了细胞分裂和凋亡之间的平衡。对癌症进展有重大影响的基因被称为癌症驱动基因,根据它们在癌症进展中的作用可分为肿瘤抑制基因(TSGs)和原癌基因(OGs)。驱动基因的发现对于癌症的预防、诊断和治疗意义重大。
目前,新驱动基因的发现主要依赖于CRISPR-Cas9系统在细胞系模型中的筛选。然而,细胞系与原代细胞差异巨大,CRISPR筛选出的基因很可能在生理学上与肿瘤相关性不大。并且尽管大多数已知的驱动基因都是基于基因变异来确定的,但证据表明,单凭基因变异不足以解释所有的癌症驱动基因。基于以上两点,有必要根据患者基因组学数据,预测癌症驱动基因;同时,如表观遗传改变等基因变异以外的特征,在预测肿瘤驱动基因时的巨大潜力亟待开发。
研 究 方 法
为了准确识别驱动基因,团队首先建立了两个阳性训练基因集与一个阴性训练基因集。阳性集包括来自CGC(癌基因统计)数据库的242个TSGs和240个OGs;阴性训练基因集包括与癌症无关的4,058个中性基因(NGs)。根据文献调研,团队构建了75个可能预测癌症驱动基因的特征。
这些特征分为四大类:a. 33个突变特征;b. 2个基因组特征;c. 27个表观遗传学特征,包括组蛋白修饰、启动子和基因体区甲基化特征、以及超级增强子比例等;d. 3个表型特征,包括CRISPR筛选数据、变异效应评分和基因表达Z值。根据特征组对分类贡献率的排名,最后筛选得到三个预测TSGs的特征组及五个预测OGs的特征组。
预 测 结 果
通过分析这些排名靠前的预测特征组,研究发现,多组蛋白修饰是TSGs最具预测性的特征组;而错义突变是预测OGs的首要特征组,表观遗传学特征是预测OGs的主要特征组。研究同时发现,组蛋白修饰和错义突变是TSGs和OGs共同的主要预测特征,表明TSGs和OGs共有某些特征,但它们对TSGs和OGs的预测能力贡献可能不同。
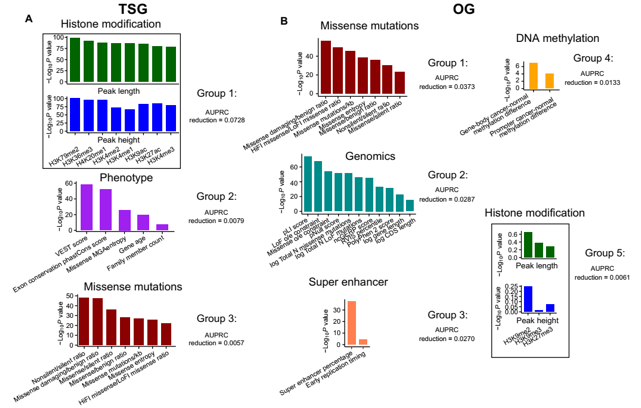
图1. TSGs与OGs预测特征组
随后,研究使用筛选后的预测特征组在基因组范围内给每个基因打分,DORGE总共预测了1172个癌症驱动基因,包括436个双功能基因。剔除掉CGC数据库中已经预测出的驱动基因后,共发现725个TSGs和515个OGs,其中537个新TSGs未包含在CancerMine或TSGene数据库中,306个新的OGs未包含在CancerMine或ONGene数据库中。

图2. DORGE预测癌症驱动基因结果与其他预测算法结果比较
在生物医学文献中搜索DORGE排名前15位的全新预测的TSGs和OGs,发现10个TSGs和12个OGs分别具有抑癌和致癌功能。特别值得注意的是,几乎所有排名靠前的TSGs都有宽的H3K4me3峰,并且大多数排名靠前的OGs在基因体区存在超甲基化。

图3. 预测排名靠前的驱动基因特征富集
研究团队进一步比较了DORGE和10种现有的基于CGC基因的癌症驱动基因预测算法,这些算法使用了四种精确性度量:敏感性(Sn)、特异性(Sp)、精确性和总体准确度。研究发现,除Sp外,DORGE在所有这些度量中表现最好(在Sp方面DORGE为0.997,最佳算法20/20+为1.000)。DORGE的优势在Sn中最为明显,其最高表现(0.611),其次是OncodriveFM(0.338)、MuSIC(0.331)和MutPanning(0.318)。结果表明,DORGE在改进现有算法的癌症驱动基因预测方面取得了重大进展。

图4. DORGE与其他预测算法性能比较
对新的TSGs和OGs进行KEGG通路分析,如预期的一样,新预测的TSGs富含TSG相关通路,如“凋亡”和“粘着斑”,新预测的OGs富含OG相关途径,如“细胞周期”和“TGF信号通路”。然而,如果忽略表观遗传学特征,DORGE预测的新TSG和OGs便不再富含某些TSG相关和OG相关的途径,这些结果再次表明表观遗传学特征对发现新的癌症驱动基因做出了独特的贡献。
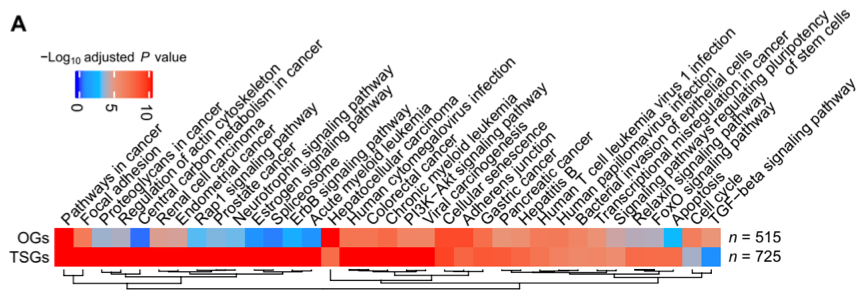
图5. 预测排名靠前的驱动基因KEGG通路富集
预测到的驱动基因的外显子和非编码区都比NGs更保守。在TSGs和OGs之间,TSGs在外显子上更加保守,而OGs在非编码区更加保守。分析结果表明,大量癌症驱动基因可能比之前认为的起源时间更早,在多细胞生物出现之前便已经存在。
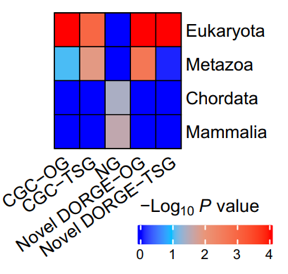
图6. 驱动基因起源富集图
最后,团队探究了TSGs和OGs表达的蛋白质与其他基因/蛋白质的相互作用情况。结果显示,TSGs和OGs往往处在蛋白互作的网络的节点位置。说明肿瘤驱动基因的改变将对整个互作网络产生重大影响。紧密联系的基因倾向于形成模块,而肿瘤驱动基因模块可以引发癌症特征,并赋予癌细胞显著的增殖优势。

图7. 驱动基因表达蛋白互作网络
研 究 小 结
李蔚教授团队首创性开发了一种机器学习工具——DORGE,通过整合遗传和表观遗传特征来识别癌症驱动基因。尽管进一步的研究还需要实验验证,但对算法预测结果的评估证实了DORGE预测未知癌症驱动基因的能力,加深了对表观遗传学在肿瘤发生过程中机制的理解,为更多潜在治疗靶点的发现及应用奠定了坚实基础。
李蔚教授
求臻医学联合创始人&首席科学家
美国加州大学(尔湾)终身教授,讲席教授
主要研究方向:设计和应用生物信息学算法来评估肿瘤等疾病发生发展过程中的全局调控机制,在大规模基因组数据及表观遗传学数据分析方面有着坚实的基础。参与完成人类基因组计划,主导完成了中国第一个微生物基因组计划等重大项目。所开发的BSMAP、RSeqQC等算法,目前已成为Bisulfite-seq、RNA-seq数据分析等多个生信领域的标准算法。现已在高影响因子期刊上发表超过190篇论文,其中23篇作为通讯作者发表在Nature、Science、Cell系列刊物(包括子刊),H-index 高达80(发表的论文中有80篇引用超过80次)。
求臻医学首席科学家李蔚教授研究团队
诚招博士后2名
研究方向
通过表观遗传的大规模数据挖掘来解释肿瘤等人类复杂疾病
岗位要求
1.获得与生物信息学相关的博士学位;
2.具有分子生物学、肿瘤学研究背景,从事过肿瘤表观遗传学工作的人员优先;
3.博士期间,以第一作者发表过涉及生物信息分析内容的SCI论文;
4.具有良好的团队合作与沟通能力,以及较强的中英文写作能力。
工作地点
求臻医学科技(北京)有限公司或美国加州大学
简历投递
Wei.li@uci.edu
目前,李蔚教授已有6位学生在美国一流研究型大学(包含哈佛医学院、匹兹堡大学和梅奥诊所),获得独立PI的教授职位,2位学生已获得中国国家青年千人学术头衔。详情可参见:https://sites.uci.edu/weililab/。







