臻话RNA | 第五期

近年来,肿瘤精准医疗飞速发展,尤其是ALK、ROS1、RET、NTRK、FGFR等以结构变异为主的靶向药物陆续上市,越来越多患者从中获益。目前,基于DNA变异分析为主的精准医疗正在被临床广泛应用,实际上,基于RNA的NGS检测(RNA-NGS)在结构变异检测上展现出更加优越的性能,已被《NCCN 非小细胞肺癌临床实践指南》、《NCCN 结肠癌临床实践指南》、《骨与软组织肿瘤二代测序中国专家共识》等指南及专家共识收录。同时,RNA种类繁多、功能复杂,RNA-NGS应用也更为广泛……上期我们了解到“RNA+分子分型:引领膀胱癌精准诊治新格局”,本期,求小臻将带领大家一起对肿瘤免疫治疗寻根探源,揭开肿瘤免疫微环境的神秘面纱。
免疫治疗已在多种肿瘤中取得令人鼓舞的结果,基于PD-L1表达、TMB、MSI均已有获批的免疫检查点抑制剂,然而,这几个生物标志物仍有其力不能及之处:肺癌PD-L1高表达人群免疫治疗获益率约40%,PD-L1表达阴性的人群仍有约10%可从免疫治疗中获益;MSI-H人群比例低,即使是结直肠癌这种具有较高概率发生MSI-H的癌种,这部分人群也不足20%;TMB除不同癌种差异较大外,也存在与MSI-H相同的情况,TMB-H人群占比较低,不足30%等。
肿瘤免疫微环境(TIME)是肿瘤免疫治疗最根本、最重要的影响因素。近年来,随着相关研究的发现及检测技术的进步,肿瘤免疫微环境逐渐走进临床科研和临床实践的视野,在免疫治疗疗效预测、预后评估等方面已取得丰硕的成果。同时,基于RNA-seq的NGS技术也成为了TIME高通量分析的有效手段之一。
肿瘤免疫微环境与免疫疗法
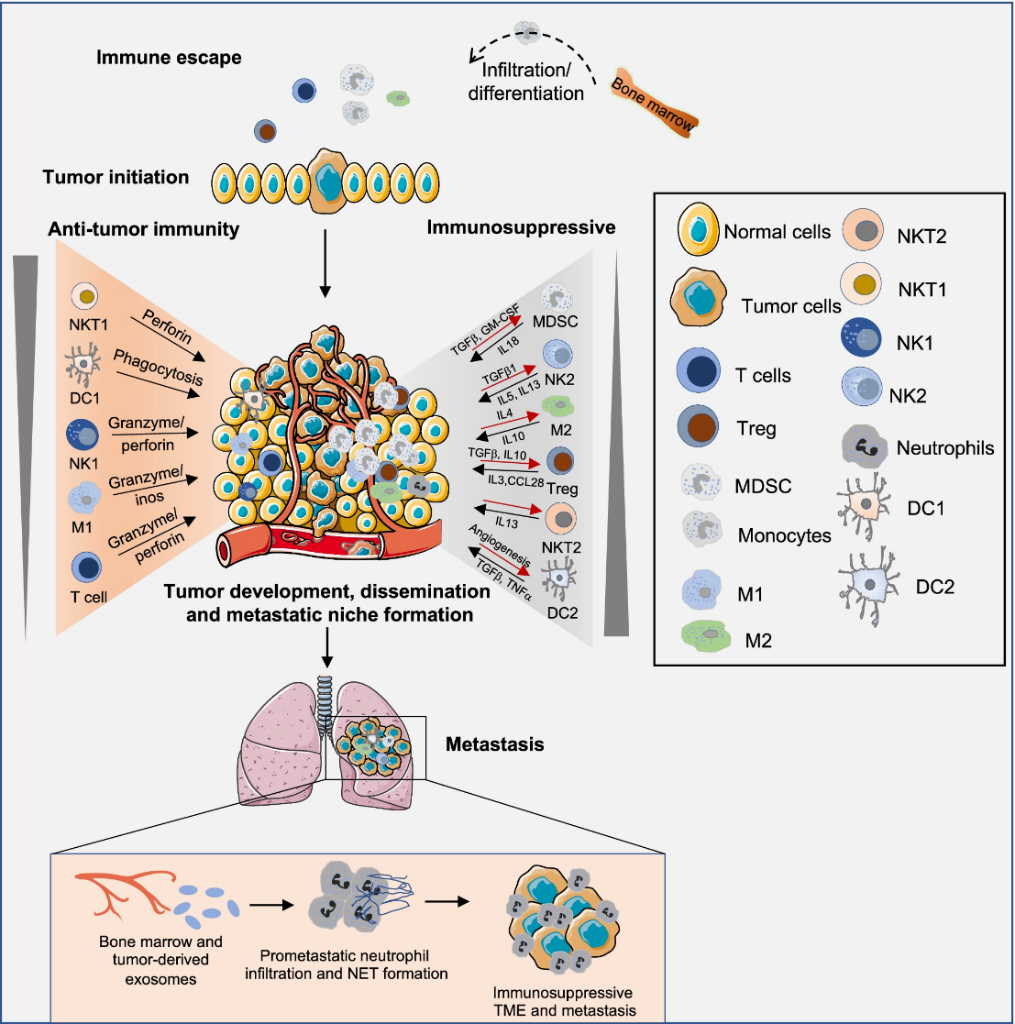
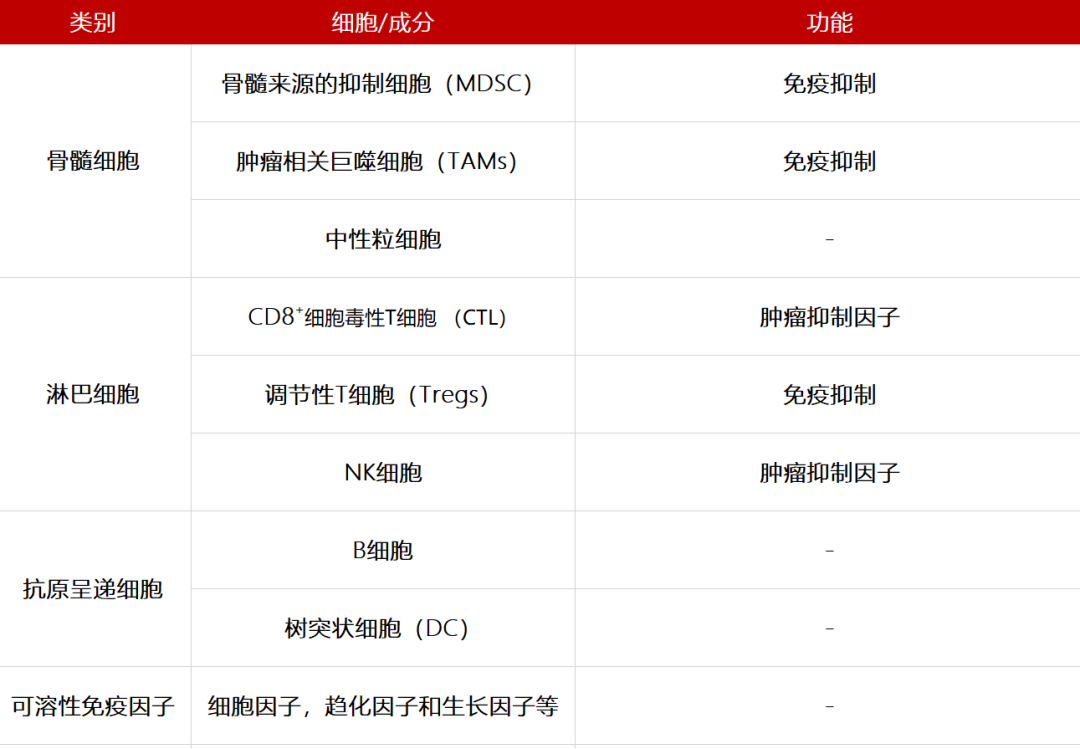
TIME 广泛分布着免疫细胞(详情见表1),TIME中免疫细胞的组成因肿瘤类型而异,并且在同一肿瘤类型的患者之间存在差异。肿瘤的发生、发展和转移受TIME控制,其中免疫抑制和抗肿瘤免疫串扰起着重要作用,如图1B所示[1]。
表1. 肿瘤免疫微环境重要组成成分[1]
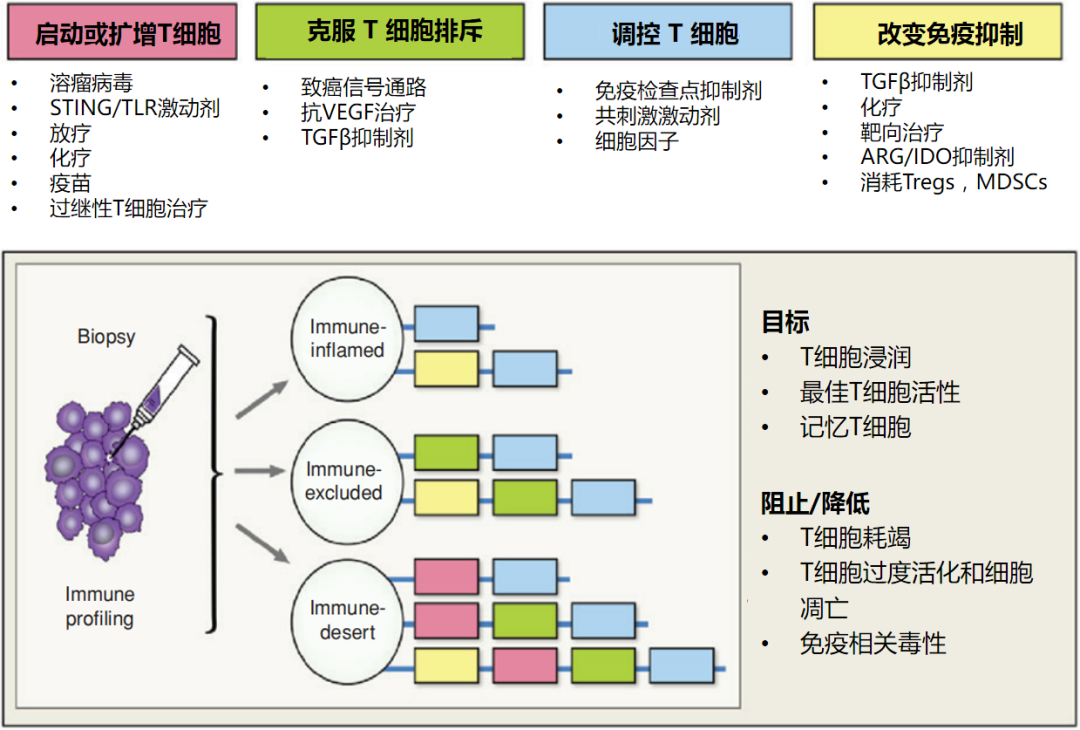
目前癌症患者的临床治疗存在两个主要障碍:一是免疫逃逸,二是获得性耐药,这也与免疫抑制微环境有关。根据免疫浸润,将肿瘤分为三种免疫亚型:免疫炎症型(Immune-inflamed)、免疫排斥型(Immune-excluded)、免疫荒漠型(Immune-desert)。其中,CD8+T细胞富集是免疫炎症型特征之一,该亚型免疫治疗获益概率高。非炎症型肿瘤(免疫排斥型和免疫荒漠型) 极少携带CD8+T细胞,通常表达与免疫抑制或耐受相关的细胞因子,包括调节性T细胞(Treg)、髓系抑制性细胞等。因此,通过免疫表型相关特征分析,可预测其免疫微环境状态,从而筛选合适的治疗组合方案,促进T细胞浸润和功能优化(图1 A)[2]。
图1A 促进T细胞浸润和功能优化的组合策略示例,B 肿瘤发展过程中的TIME串扰[1]
(左右滑动查看更多)
RNA-seq在肿瘤免疫微环境检测中的应用
RNA-seq提供了一种有效的高通量技术可靠地表征肿瘤免疫微环境(TIME)。基于该技术可以揭示抗肿瘤免疫反应中免疫浸润组成、表达表型及免疫组库等关键组成部分(见图2)[3]。
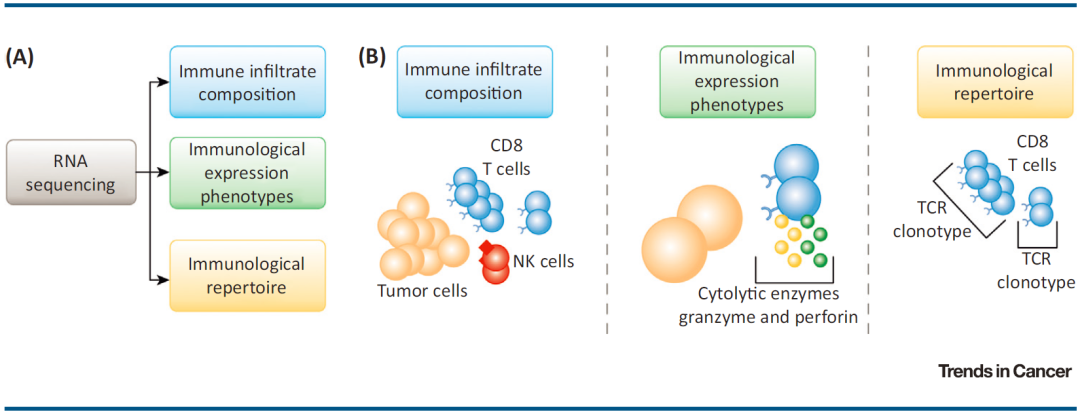
图2A RNA测序揭示抗肿瘤免疫反应的关键组成部分 B RNA 表达数据的应用
2.1 表征肿瘤免疫微环境的RNA分子特征已展现出重要的疗效预测价值
Ayers等人经过严格的多步骤验证过程,筛选18个T细胞炎性基因,对这些基因表达量的内参标准化值采取加权总和计算T细胞炎性基因表达谱评分(GEP score,也有报道称为TIS score)。
GEP score在头颈鳞癌、胃癌、黑色素瘤等多种实体瘤中预测了帕博利珠单抗治疗的反应,可对响应人群和无响应人群进行分层,并且其与TMB具有较低相关性(r = 0.221, P < 0.05),TMBhi且GEPhi的人群具有更好的PFS也证明了这一结论,这也表明TMB与GEP score联合使用将有助于筛选最佳获益人群[4,5]。
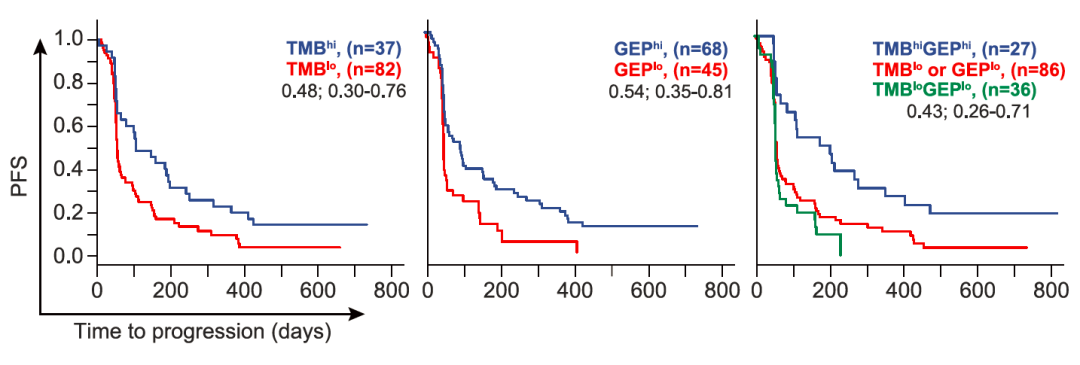
图3.泛癌种患者队列抗PD-1治疗后TMB、GEP评分与PFS的关系[5]
在针对TCGA来源的多癌种RNAseq数据分析研究中,也得出了TIS score与TMB的相关性较低的结论。具有较高中位TIS score的癌种,如晚期或转移性肾透明细胞癌、黑色素瘤、肺癌、头颈肿瘤,已取得抗PD-L1治疗临床获益。而肾癌(图4中标红)对免疫检查点抑制剂有响应人群可通过TIS score进行预测,但不能通过TMB进行较好的预测[6]。
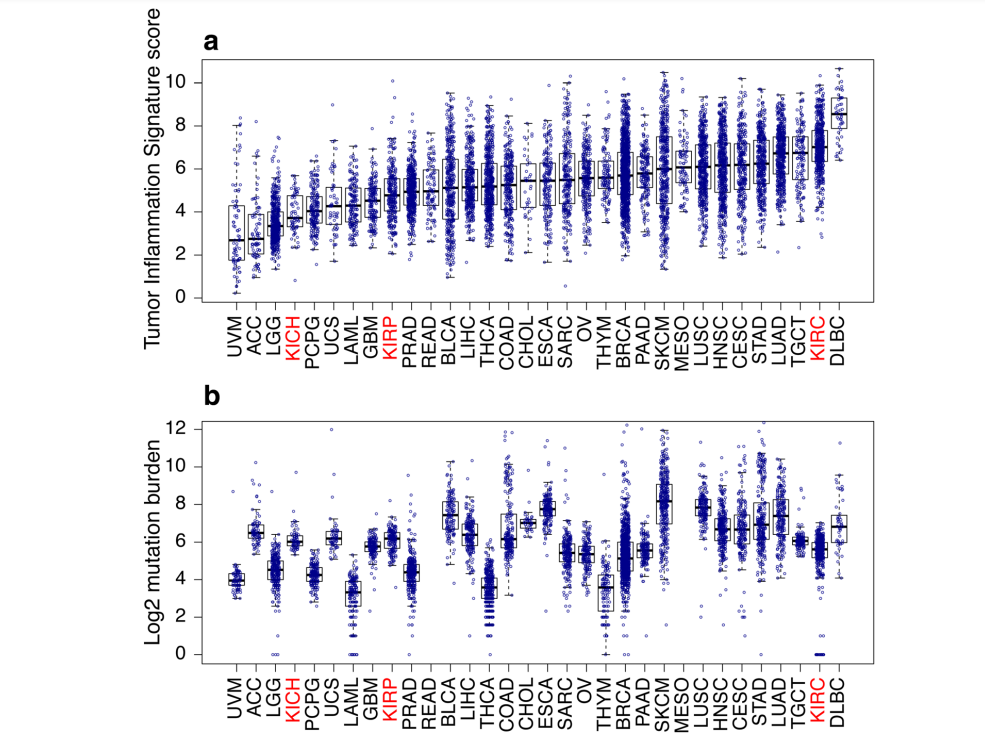
图4. 所有患者的TIS score(数据来自TCGA)
2.2 基于转录组分析免疫浸润及表达表型,反映肿瘤免疫反应的关键特征
早期发表在Cell上的研究就发现,通过分析免疫细胞溶解活性(CYT)水平,可以反映CD8+T细胞的状态以及患者抗PD-L1和抗CTLA-4治疗的临床反应[7]。通过PD1、CD44和其他表型标记物的表达以及转录因子T-bet和EOMES的表达来识别耗竭态的CD8+T细胞的亚群, 耗竭的T细胞失去强大的效应器功能[8]。
多项临床研究表明,TIME特别是关键效应细胞(如CD8 T细胞)的存在与否,可影响肿瘤免疫应答[9,10,11]。RNAseq一次性检测了所有转录基因的表达数据,可以对表达数据进行复杂的分析,以确定 CD8 T 细胞和其他免疫细胞类型存在与否以及相对丰度。
目前预测免疫浸润的大多数方法使用反卷积方法或基因集富集分析 (GSEA) 方法。前者的主要优势在于可以输出每种免疫细胞类型的相对比例,不足是其性能受参考表达谱的影响,代表性分析方法为CIBERSORT。后者根据精选基因的表达排名计算富集评分,优点是它们不需要参考表达谱,可以独立分析免疫细胞,避免了反卷积方法发现的多重共线性问题,同时,GSEA方法因为基于基因表达的等级,而不是实际的基因表达值,也可能对批次效应更可靠;不足是富集分数不一定与实际丰度呈线性关系[1]。
对不同免疫表型特征的分析可能通过与TIS结合来进一步解剖免疫反应。
2.3 RNA-seq用于解析免疫组库
由于可以捕获表达和序列信息,RNA-seq还可用于解析免疫组库。研究表明,T细胞受体(TCR)和B细胞受体(BCR)库可能是与有效免疫治疗反应相关的预测性生物标志物[12,13,14]。虽然也可以使用DNA测序进行相关分析,但由于只有重组受体表达,RNAseq的背景噪声较少,相对而言具有一定的优势。
总结
免疫微环境作为非常有潜力的生物标志物之一,已受到临床广泛的关注。探索与反应和耐药相关的肿瘤微环境将有助于更精确地识别从免疫治疗中获益的人群。同时,RNA-seq以高通量方式获取临床样品中全转录组表达数据和序列信息,能够为肿瘤免疫微环境提供全面的分析。期待在基于RNA的肿瘤免疫微环境检测的加持下,更多患者能够获益于免疫治疗。

求臻医学ChosenMaster
肿瘤免疫微环境检测方案
求臻医学基于RNA测序技术开发了ChosenMaster肿瘤免疫微环境检测方案,通过评估不同基因表达量,进行T细胞炎性基因表达谱评分(GEP score)(涵盖18基因)、六种免疫表型相关特征(涵盖350+基因)分析。该检测方案采用去核糖体链特异性建库方式,可有效弥补FFPE等降解程度较高的样本5’端RNA信息丢失的问题,极大地提升了检测准确性。
同时,该检测方案整合来自TCGA的13类癌种RNAseq数据集作为背景集,可直观地观察检测结果与其对应癌种背景集的关系,评估实体瘤肿瘤免疫微环境状态,预测免疫疗效,为肿瘤免疫治疗临床实践提供参考。