近日,中国医科大学附属盛京医院第六普通外科(肝胆外科移植外科)刘宝林教授与求臻医学团队合作,开发一种新型多基因预后模型,可有效预测结直肠癌患者的预后生存情况,为结直肠癌患者预后及治疗的分层管理提供指导依据。该模型基于结直肠癌基因表达数据,利用统计学算法筛选出具有潜在预后价值的9-基因特征,组合构建新型多基因预后模型,并于多组数据集中验证了该模型的预后价值。目前相关研究成果发表于Frontiers in Oncology(IF=5.738,中科院二区)。

结直肠癌(CRC)是目前我国发病率及死亡率最高的恶性肿瘤之一。随着精准医疗理念的深入及肿瘤相关分子标志物的研究进展,基于患者病理分期(TNM)、年龄或影像学等传统指标进行患者预后评估无法满足临床实际需求,迫切需要探寻新型预后分子标志物,进而指导结直肠癌患者术后管理以及精准治疗方案的制定。近年来,越来越多的研究发现癌症的发生发展是由于调控某些关键癌症基因的RNA失调导致。因此,本文基于结直肠癌RNA-Seq数据,利用统计学方法筛选基因特征,进而组合构建出一个有效的多基因预后模型,来准确预测结直肠癌患者的预后生存情况。
首先,本研究使用单因素Cox回归、Lasso回归和多变量Cox回归分析,从癌症基因组图谱(TCGA)数据库的结直肠癌RNA测序数据中筛选出与结直肠癌患者预后显著相关的基因。然后,根据9个候选基因的表达情况计算出每个病人的风险分数(RS),将患者分为高低风险组,从而预测患者的预后情况。
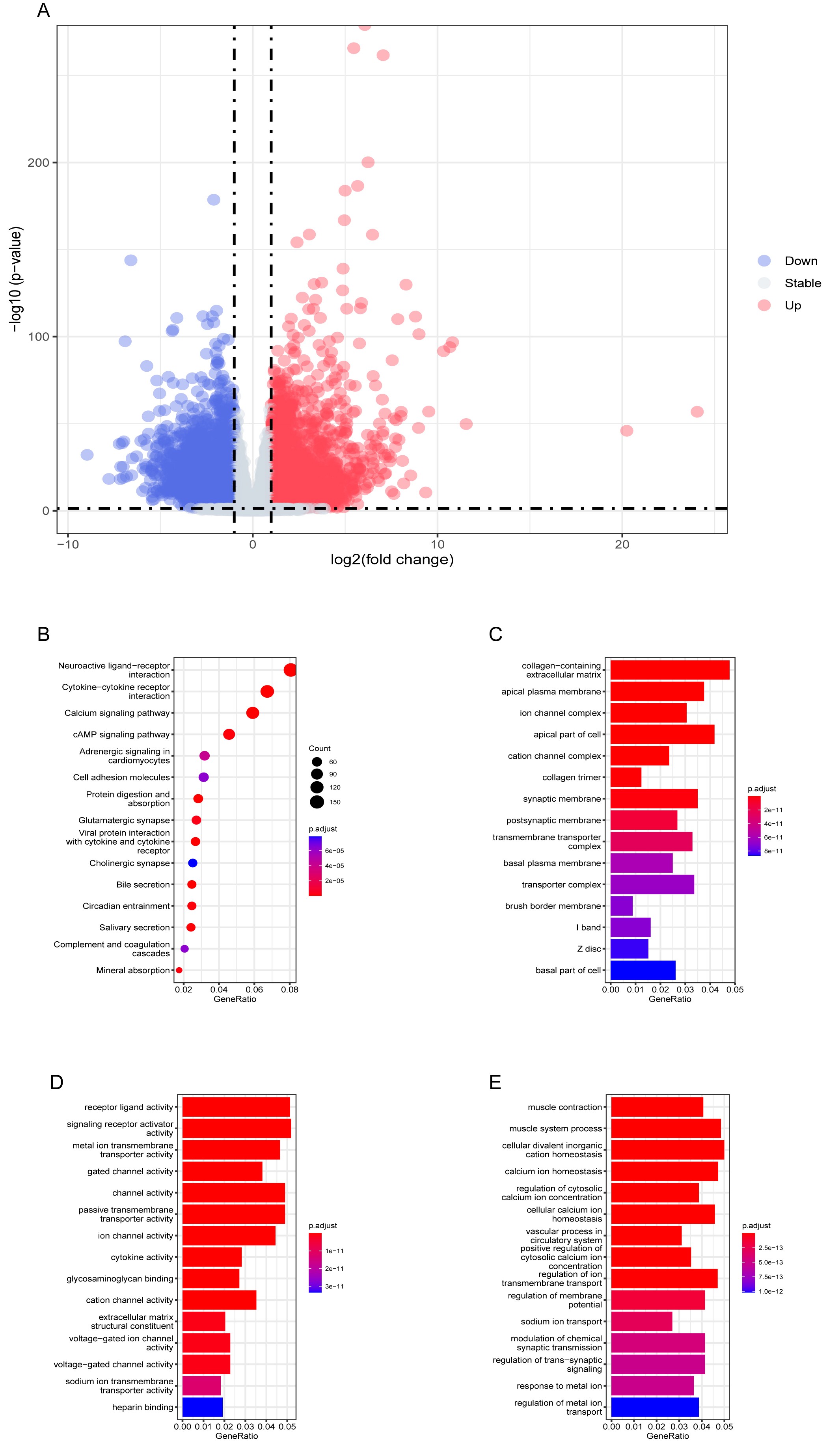
首先在训练数据集中分析得到结直肠癌肿瘤组织和癌旁组织之间的5286个显著差异表达基因,并对其进行富集分析。
再通过使用单因素Cox回归、Lasso回归和多变量Cox回归分析进一步筛选,得到9个与结直肠癌患者的预后高度相关的基因,根据这9个基因的表达谱构建预后模型,对患者进行风险评估。结果显示被分为高风险组的患者(红色)死亡的比例显著高于低风险组。
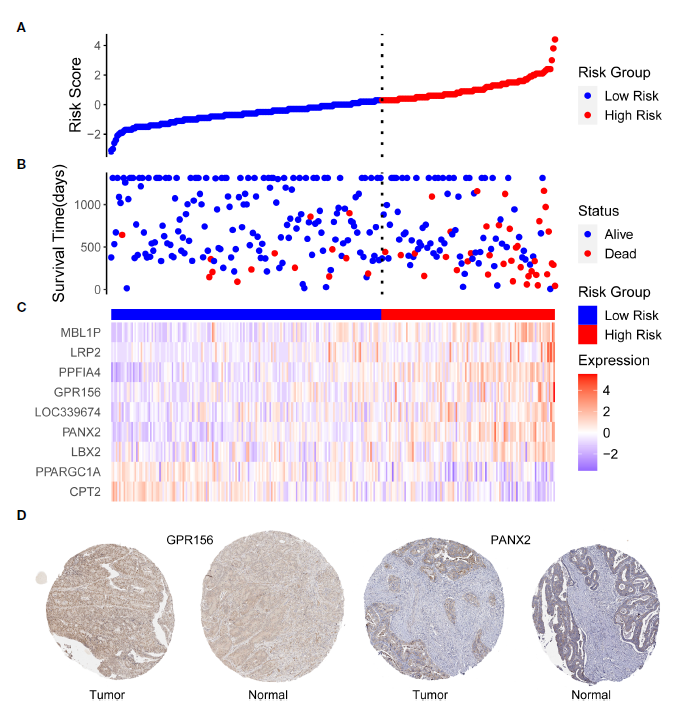
图2. TCGA患者使用预后模型分组情况,模型基因表达量热图以及在肿瘤和正常样本中表达情况
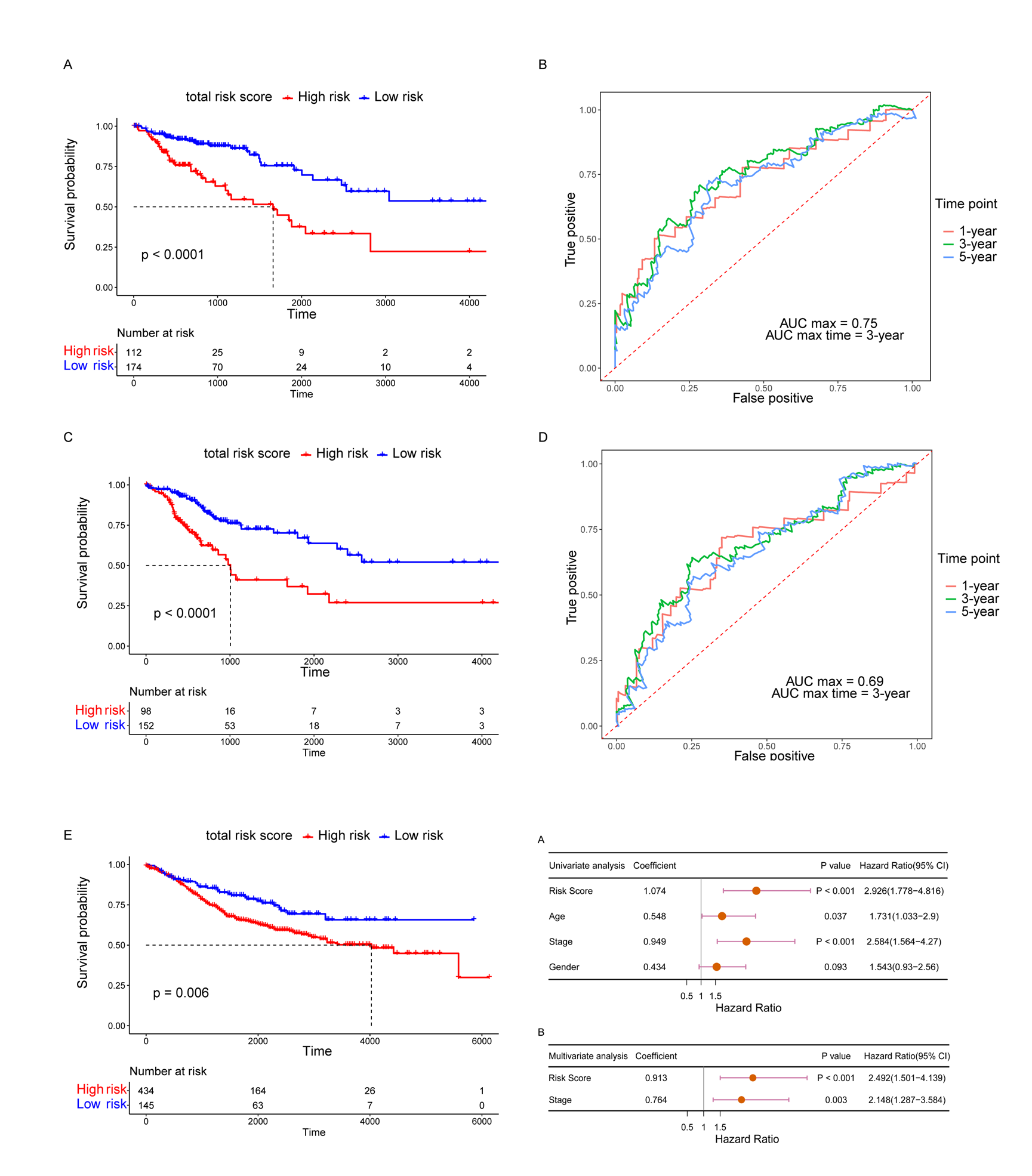
使用预后模型对训练数据集进行K-M曲线分析,高、低风险组的总体生存(OS,P<0.0001)和无疾病生存期(DFS,P<0.0001)存在显著差异,该模型预测AUC值达到0.75。结果表明本文构建的预后模型可以以较高的准确性有效的预测TCGA数据集中结直肠癌患者的预后情况。之后选择在TCGA验证数据集和三个GEO独立数据集中对预后模型的预测效能进行验证。结果表明该预测模型在所有验证集中都表现出了良好的预测能力,说明该模型不仅可以准确地预测结直肠癌患者的预后情况,同时还具有广泛的适用性。最后,本研究将提出的多基因预后模型作为患者的预后指标之一,与其他传统临床特征(年龄,分期)进行比较,结果表明多基因模型预测效能要明显优于传统指标。
本研究表明多基因模型可以有效地预测结直肠癌患者的预后,精准地评估患者预后风险,可为结直肠癌患者预后及治疗的分层管理提供指导依据。同时本研究也为结直肠癌基础实验研究提供了一个重要的研究方向。
主任医师
中国医科大学附属盛京医院
第六普通外科 肝胆外科移植外科
•医学博士 留美博士后 博士研究生导师
•中华医学会外科分会 会员
•美国外科移植协会 会员
•国际肝胆胰协会中国分会 委员
•中华医学会东北三省肠外肠内营养支持专业委员会 常委
•沈阳市医学会肛肠分会 常委
•辽宁省中西医结合协会肛肠分会 常委
•中国北方微创手术协作中心 委员
•辽宁省医学会外科分会结直肠外科学组 委员
•中国非公立医疗机构协会胆石病专业委员会 常委
•辽宁省器官移植学分会 委员
•中国抗癌协会营养与支持治疗专业委员会加速康复学组 常委
•中国医药教育协会医疗器械管理专业委员会 常委
•中国医药教育协会医疗器械管理专业委员会肝胆器械学组 副主任委员
•中国医师协会结直肠肿瘤专业委员会结直肠肝转移专业委员会 委员
•辽宁省生命科学协会肝胆胰专科委员会 委员
•世界内镜医师协会肝胆胰肠协会 副主任委员
•辽宁省医学会外科分会器官移植学组 委员
•辽宁省器官移植学分会 常委
•辽宁省细胞生物学会先进医工融合技术专委会 常委
参考文献:
1. Bray F, Ferlay J, Soerjomataram I, Siegel RL, Torre LA, Jemal A. Global Cancer Statistics 2018: Globocan Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries. CA: a cancer journal for clinicians (2018) 68(6):394-424. Epub 2018/09/13. doi: 10.3322/caac.21492.
2. Ferlay J, Steliarova-Foucher E, Lortet-Tieulent J, Rosso S, Coebergh JW, Comber H, et al. Cancer Incidence and Mortality Patterns in Europe: Estimates for 40 Countries in 2012. European journal of cancer (Oxford, England : 1990) (2013) 49(6):1374-403. Epub 2013/03/15. doi: 10.1016/j.ejca.2012.12.027.
3. Galon J, Costes A, Sanchez-Cabo F, Kirilovsky A, Mlecnik B, Lagorce-Pagès C, et al. Type, Density, and Location of Immune Cells within Human Colorectal Tumors Predict Clinical Outcome. Science (New York, NY) (2006) 313(5795):1960-4. Epub 2006/09/30. doi: 10.1126/science.1129139.
4. Goodall GJ, Wickramasinghe VO. Rna in Cancer. Nature reviews Cancer (2021) 21(1):22-36. Epub 2020/10/22. doi: 10.1038/s41568-020-00306-0.
5. Cooper TA, Wan L, Dreyfuss G. Rna and Disease. Cell (2009) 136(4):777-93. Epub 2009/02/26. doi: 10.1016/j.cell.2009.02.011.
6. Wahab R, Gopalan V, Islam F, Mamoori A, Lee KT, Lu CT, et al. Expression of Gaec1 Mrna and Protein and Its Association with Clinical and Pathological Parameters of Patients with Colorectal Adenocarcinoma. Experimental and molecular pathology (2018) 104(1):71-5. Epub 2018/01/18. doi: 10.1016/j.yexmp.2018.01.004.
7. El-Ashmawy NE, Al-Ashmawy GM, Hamouda SM. Long Non-Coding Rna Fam83h-As1 as an Emerging Marker for Diagnosis, Prognosis and Therapeutic Targeting of Cancer. Cell biochemistry and function (2021) 39(3):350-6. Epub 2020/11/08. doi: 10.1002/cbf.3601.
8. Zhao J, Guo C, Ma Z, Liu H, Yang C, Li S. Identification of a Novel Gene Expression Signature Associated with Overall Survival in Patients with Lung Adenocarcinoma: A Comprehensive Analysis Based on Tcga and Geo Databases. Lung cancer (Amsterdam, Netherlands) (2020) 149:90-6. Epub 2020/10/02. doi: 10.1016/j.lungcan.2020.09.014.
9. Ma B, Li Y, Ren Y. Identification of a 6-Lncrna Prognostic Signature Based on Microarray Re-Annotation in Gastric Cancer. Cancer medicine (2020) 9(1):335-49. Epub 2019/11/20. doi: 10.1002/cam4.2621.